Équipe : Mucines, cancer et résistance aux drogues
Notre équipe « Mucines, cancer et résistance aux drogues » est composée de chercheurs, d’enseignants-chercheurs, de chercheurs hospitalo-universitaires cliniciens, biologistes et anatomopathologistes, d’ingénieurs, de techniciens et d’étudiants.
Nous projet porte sur le rôle des mucines dans le cancer et sur les mécanismes de résistance des tumeurs aux traitements chimiothérapeutiques. Les tumeurs solides étant caractérisées par une importante hétérogénéité au niveau intra-tumoral et dans leur environnement (présence de différentes matrices, de différents types cellulaires : cellules stromales, immunitaires, inflammatoires,…), nous travaillons à mieux comprendre (i) les mécanismes impliqués dans la chimiorésistance (signalisation intracellulaire, épigénétique, métabolisme,…), (ii) l’impact des interactions tumeur-stroma sur le comportement de la tumeur et (iii) l’hétérogénéité tumorale (cartographie et profilage moléculaires, approches cellule unique et multi-omiques).
Pour cela, nous développons une recherche fondamentale, pré-clinique et translationnelle en cancérologie en collaboration avec les services hospitaliers du CHU de Lille avec le but de transférer nos données en pratique clinique (nouveaux marqueurs, nouvelles cibles thérapeutiques, nouveaux protocoles, médecine personnalisée) afin d’améliorer la prise en charge et le traitement des patients (accès aux cohortes régionales de patients et aux banques de données clinico-biologiques nationales).
Nous développons aussi une recherche interdisciplinaire avec les chimistes (nouvelles drogues, chimie thérapeutique), avec les physiciens (modèles microfluidiques 4D, modèles organe/tissu-sur-puce) et avec les bioinformaticiens (intelligence artificielle pour le cancer).
Ces travaux ont pour finalité de découvrir de nouveaux marqueurs phénotypiques, pronostiques, prédictifs, de réponse aux traitements (orientations des traitements) dans les cancers oeso-gastriques, coliques, pancréatiques et rénaux et aussi d’identifier de nouvelles cibles thérapeutiques (amélioration de l’efficacité des traitements) afin de proposer de nouveaux outils aux cliniciens pour mieux prendre en charge et mieux traiter le patient (médecine personnalisée).
Nous accueillons chaque année entre 10 et 15 étudiants (scientifiques et médecins) en M1, Master 2 Recherche, des doctorants et des chercheurs post-doctoraux. Nous sommes affiliés à l’école doctorale biologie-santé de Lille (http://edbsl.univ-lille2.fr/).
Notre équipe est une équipe phare de la dynamique régionale en cancérologie soutenue par l’Inserm (https://www.inserm.fr/), le CNRS (http://www.cnrs.fr), la région Hauts de France (https://www.hautsdefrance.fr/), l’institut ONCOLille (https://oncolille.univ-lille.fr/), le cancéropôle Nord-Ouest (https://www.canceropole-nordouest.org/), l’université de Lille (https://www.univ-lille.fr/), le Centre Hospitalier Universitaire de Lille (CHU) (https://www.chu-lille.fr/) et le Centre régional de lutte contre le cancer Oscar Lambret (COL) (https://www.centreoscarlambret.fr/).
Nos thèmes de recherche sont :
A- Rôle des mucines MUC1 et MUC4 dans la cancérogenèse épithéliale et la chimiorésistance
A.1- Rôle des mucines MUC1 et MUC4 dans les cancérogenèses pancréatique, oesophagienne, gastrique, colique et rénale. Modèles in vitro (modèles cellulaires 2D, 3D, organoïdes et 4D microfludiques (collaboration avec les physiciens), in vivo (modèles murins transgéniques, xénogreffes de cellules et PDX) et ex vivo (échantillons de patients, cohortes).
A.2- Étude de la relation structure-fonction du complexe MUC4-ErbB2 et du site de clivage de MUC1-CT. Ciblage thérapeutique et découverte de nouveaux candidats-médicaments (collaboration avec les chimistes).
B- Étude des mécanismes moléculaires responsables de la résistance/récidive des tumeurs
B.1- Compréhension des mécanismes de résistance aux chimiothérapies et d’échappement aux drogues (dormance tumorale, reprogrammation cellulaire). Plasticité épigénétique des cellules cancéreuses (méthylation ADN, code histones, ARN non codants, métabolisme, état souche).
B.2- Recherche transversale/translationnelle avec l’hôpital (cohortes, BCB) : Recherche de facteurs/marqueurs pronostiques (identification et classification des tumeurs) et prédictifs (orientation thérapeutique et prise en charge des patients). Comparaison tumeur primaire/récidive/métastase. Hétérogénéité de la tumeur (cartographie moléculaire de la tumeur, approches cellule unique et multi-omiques). Utilisation de l’intelligence artificielle (machine/deep learning, collaboration avec les bioinformaticiens).
> PERSONNEL CHERCHEURS
> PERSONNEL INGÉNIEUR/TECHNICIEN
> ÉTUDIANTS
 | Dr Isabelle VAN SEUNINGEN, chercheur DRCE CNRS, PhD |
 | Dr Vincent SENEZ, chercheur DR1 CNRS, PhD vincent.senez(@)univ-lille.fr |
 | Dr Nicolas JONCKHEERE, chercheur CRCN Inserm, PhD |
 | Dr Bernadette NEVE, chercheur CRCN Inserm, PhD |
 | Dr Audrey VINCENT, chercheur CRCN Inserm, PhD Responsabilité collective : Responsable de la plate-forme de culture cellulaire 3D OrgaRES, ORGALille et OrgaNO |
 | Pr Michaël PERRAIS, Professeur des Universités PU, PhD |
 | Pr Emmanuelle LETEURTRE, anatomopathologiste, MD PhD |
 | Pr Marie-Pierre BUISINE, biologiste, PharmD PhD |
 | Pr Guillaume PIESSEN, chirurgien digestif, MD PhD |
 | Pr Sébastien AUBERT, anatomopathologiste, MD PhD |
 | Pr Viviane GNEMMI, anatomopathologiste, MD PhD |
 | Dr Julie LECLERC, biologiste, PharmD PhD |
 | Dr Lucie COPPIN, biologiste, PharmD PhD |
 | Dr Anne-Frédérique DESSEIN, biologiste, PharmD PhD |
 | Pr Stéphanie TRUANT, chirurgien digestif, MD PhD |
 | Pr Pascal PIGNY, biologiste, PharmD PhD |
 | Dr Julie VEZIANT, chirurgien digestif, MD PhD julie.veziant(@)chu-lille.fr |
 | Dr Clarisse EVENO, Chirurgien, MD PhD |
 | Dr Mehdi EL AMRANI, Chirurgien, MD PhD |
 | Dr Jean-Baptiste GIBIER, anatomopathologiste, MD PhD Recherche : Rôle de MUC1 au cours de l’insuffisance rénale aiguë et chronique. Étude de MUC1 dans la réponse inflammatoire après stress rénal. Étude des mécanismes conduisant à la fibrose rénale. |
| Dr Arnaud JANNIN, Endocrinologue, MD PhD Recherche : Étude de la résistance aux traitements dans les tumeurs neuroendocrines pancréatiques. Mots-clés : métabolisme, données -omiques, tumeurs neuroendocrine pancréatiques | |
| Dr Anthony TURPIN, Oncologue, MD PhD Recherche : Étude de la résistance aux traitements dans les tumeurs pancréatiques. Mots-clés : résistance, protocole thérapeutique, données -omiques, tumeurs pancréatiques et digestives |
PERSONNEL INGÉNIEUR/TECHNICIEN
 | Belinda DUCHÊNE, Assistante Ingénieure Inserm belinda.duchene(@)inserm.fr |
 | Dr Mouloud SOUIDI, PhD, Ingénieur d’étude Université de Lille mouloud.souidi(@)inserm.fr |
 | Dr Sonia PAGET, PhD, Ingénieur d’étude CDD U. Lille Responsable technique plateforme OrgaRES Compétences : biologie moléculaire et cellulaire, modèles cellulaires 3D organoïdes, technologie Crispr-Cas9, réglementation produits biologiques sonia.paget(@)inserm.fr |
Tristan GIRGIN (D2 doctorant) tristan.girgin(@)etu.univ-lille.fr | |
Felix ROYER (D4 doctorant) Allocation doctorale I-Site PEARL – Marie Sklodowska-Curie COFUND felix.royer.etu(@)univ.lille.fr | |
Robin HOUSSIER (D3 Doctorant) robin.houssier(@)student.junia.com | |
Arnaud JANNIN (D3 doctorant) Mécanismes de résistance aux chimiothérapies des tumeurs neuro-endocrines pancréatiques bien différenciées arnaud.jannin(@)chu-lille.fr | |
Leila MOUM (D3 doctorante) Résolution structurale et ciblage thérapeutique du complexe oncogénique MUC4-HER2 dans le traitement du cancer du pancréas leila.moum.etu(@)univ-lille.fr | |
Emma Bourdon (D2 doctorante) Rôles de la mucine MUC1 dans les phénomènes de chimiorésistance au cisplatine dans le cancer du poumon emma.bourdon(@)inserm.fr | |
Laura PAVIOT (D2 doctorante) laura.paviot@inserm.fr |
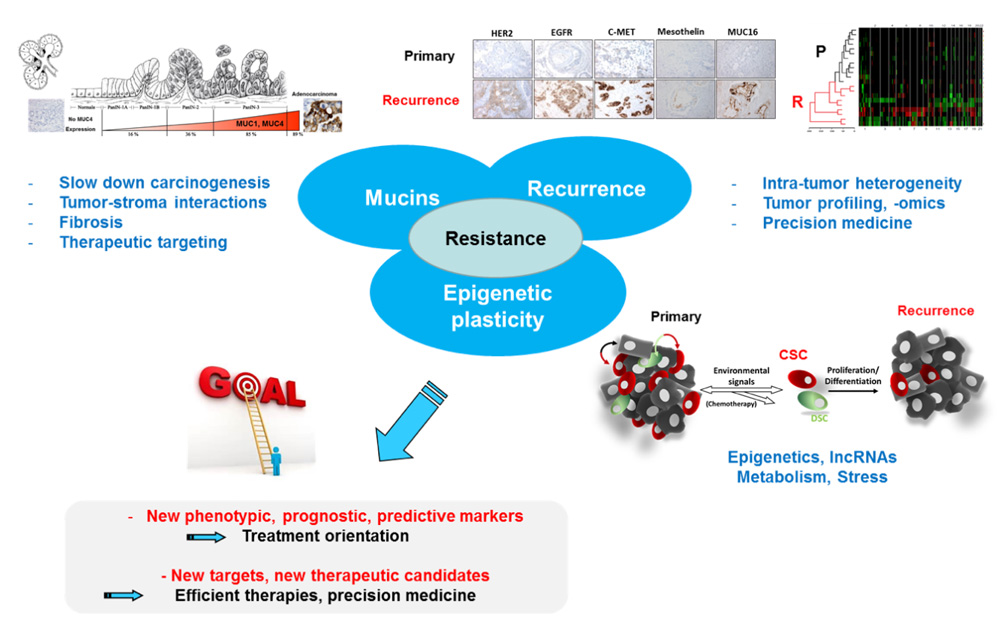
Notre stratégique scientifique et nos projets pour 2020-2024 sont basés : 1- sur la grande quantité de données obtenues lors du précédent quinquennat concernant les mécanismes de résistance de la tumeur et la découverte de biomarqueurs dans les cancers oeso-gastriques, pancréatiques, coliques et rénaux (voir liste de publications) et 2- sur la création de l’institut de recherches sur le Cancer de Lille ONCOLille, auquel CANTHER est affiliée, en 2020 sur le campus hospitalo-universitaire dont les objectifs scientifiques seront de développer une recherche fondamentale et translationnelle sur les thèmes de la résistance et de la dormance tumorales.
Notre projet d’équipe porte sur les mécanismes responsables de la résistance tumorale envers les traitements chimiothérapeutiques avec une emphase sur : 1- le rôle des mucines membranaires, 2- le rôle des mécanismes épigénétiques sur la plasticité des cellules cancéreuses et 3- la comparaison systématique du profil moléculaire de la tumeur primaire et de sa récidive (voir figure ci-dessous). Ceci devrait nous permettre (i) d’apporter des nouvelles connaissances quant à ces mécanismes et (ii) de développer de nouveaux modèles d’étude (interdisciplinarité) et de trouver de nouveaux marqueurs pronostiques/prédictifs afin de mieux prendre en charge les patients et de proposer des meilleurs traitements plus adaptés à la tumeur et à l’hôte (médecine de précision), ceci en collaboration avec les cliniciens, les pathologistes et les biologistes de l’équipe.
1- Rôles de MUC1 et MUC4 dans la chimiorésistance
Nos projets visent à mieux comprendre le rôle des mucines membranaires MUC1 et MUC4 dans la cancérogenèse et la chimiorésistance. Nous utilisons une triple approche (in vitro, in vivo, ex vivo). Nous utilisons comme modèles d’étude des cancers particulièrement résistants aux traitements (cancer du rein et cancer du pancréas) dans lesquels MUC1 et MUC4 sont surexprimées afin de mieux comprendre leurs rôles dans ces processus (Jonckheere 2017; Jonckheere 2014; Jonckheere 2010; Jonckheere 2008).
1.1 MUC1 et cancer du rein (Responsable: Michael Perrais ; Co-Investigateurs: Dr Viviane Gnemmi, Pr Sébastien Aubert, Dr Jean-Baptiste Gibier)
Dans le cancer du rein, nous avons montré que MUC1 est un acteur de la chimiorésistance aux thérapies conventionnelles et aux thérapies ciblées (Aubert 2009, Bouillez 2014, Gnemmi 2014, Gibier 2017). Dans ce projet, nous voulons décrypter les mécanismes impliqués dans la résistance via le clivage de MUC1-C. Nous utiliserons des approches de transcriptomique et de protéomique (collab. Pr I. Fournier, laboratoire PRISM, Villeneuve d’Ascq). Nous évaluerons l’effet de mutations sur la migration, l’invasion, la prolifération et la chimiorésistance des cellules cancéreuses. Nous collaborerons avec les chimistes (Pr N. Lebègue, centre LilNCog, Inserm UMR1172, Lille) pour développer une stratégie afin de bloquer le clivage de MUC1-C. En parallèle, nous développerons des nanobodies appelés anticorps VHH contre MUC1-C (Hybrigenics services, Paris). Enfin, grâce à nos modèles murins précliniques et au KO Muc1, nous étudierons la relation entre MUC1, fibrose rénale inflammatoire et développement du cancer (collab. Dr C. Cauffiez, Dr N. Pottier & Pr F. Glowacki/équipe sénescence Canther).
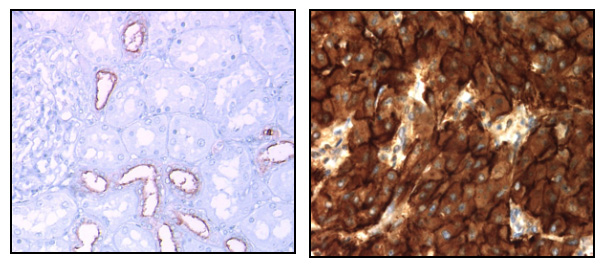 Expression de MUC1 dans le rein normal (gauche) et surexpression dans le cancer du rein métastatique (droite)
Expression de MUC1 dans le rein normal (gauche) et surexpression dans le cancer du rein métastatique (droite)
1.2. Rôle de MUC4 dans la cancérogenèse pancréatique et la résistance tumorale (responsables : Dr Nicolas Jonckheere, Dr Isabelle Van Seuningen, Co-Investigateurs: Pr Emmanuelle Leteurtre, Dr Audrey Vincent, Dr Bernadette Neve, Dr Lucie Coppin, Pr Stéphanie Truant, Dr Mehdi El Amrani)
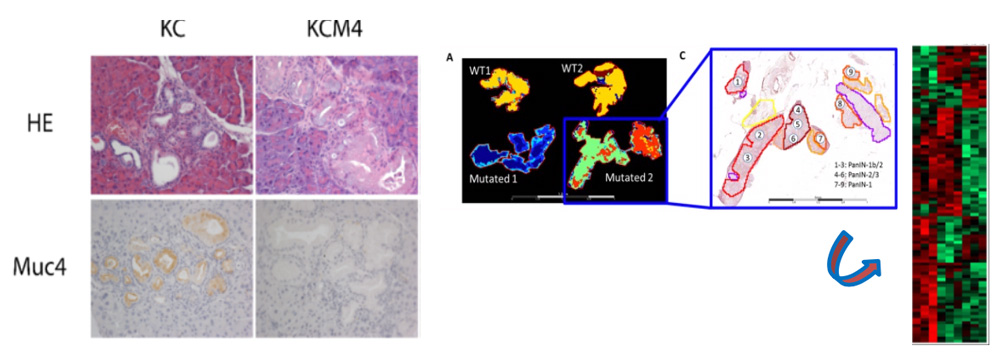 Expression de Muc4 dans le cancer du pancréas chez la souris (KC) et chez la souris cancéreuse pancréatique déficiente en Muc4 (KCM4) (gauche). Hétérogénéité moléculaire intratumorale et cartographie des PanIN par MALDI-MSI (droite)
Expression de Muc4 dans le cancer du pancréas chez la souris (KC) et chez la souris cancéreuse pancréatique déficiente en Muc4 (KCM4) (gauche). Hétérogénéité moléculaire intratumorale et cartographie des PanIN par MALDI-MSI (droite)
1.2.a. MUC4 et lésions précoces
L’adénocarcinome pancréatique (PDAC) est une maladie très complexe arborant une hétérogénéité intratumorale (HIT) importante entre les cellules tumorales, les lésions précoces de type PanINs (PanIN1a-b/2/3) et le stroma fortement fibreux. Nous avons pour objectif de comprendre le rôle de MUC4 dans l’initiation de la cancérogenèse pancréatique par la caractérisation du modèle murin préclinique triple transgénique déficient en Muc4 appelé KCM4 (Pdx1-Cre; LstopL-KrasG12D; Muc4KO). Nous établirons la cartographie moléculaire et la signature protéomique des PanIN KCM4 par l’utilisation de la technologie MALDI-MSI (collab. Pr I. Fournier, laboratoire PRISM, Villeneuve d’Ascq). Les marqueurs d’intérêt identifiés seront ensuite étudiés pour leur impact sur les propriétés des cellules cancéreuses. Avec les pathologistes, nous voulons trouver de nouveaux marqueurs de réponse en utilisant une cohorte rétrospective de « longs survivants » (CAPALONG) que nous avons mise en place en collaboration avec les centres hospitaliers inter-régionaux (Lille, Amiens, Caen, Rouen) grâce à un soutien du Cancéropôle Nord-Ouest (coordination Pr E. Leteurtre). Avec les bioinformaticiens, nous voulons développer des outils en utilisant l’intelligence artificielle (machine/deep learning) afin d’identifier les lésions précoces de manière systématique et pouvoir stratifier les patients et orienter les traitements (collab. Pr M. Elati, équipe plasticité Canther ; Dr F. Wyndal, Dr H. Benhabiles, Yncrea, Lille).
1.2.b. MUC4 et chimiorésistance
Dans ce projet, nous voulons élucider les mécanismes responsables de la résistance (innée et induite) aux drogues (gemcitabine/FOLFIRINOX) et aborder les interactions tumeur-stroma. Pour cela nous avons mis en place des modèles in vitro (2D/3D chimiorésistants) et in vivo (PDX chimiorésistants) et utiliserons des stratégies gain et perte de fonction (shARN, Crispr/cas9). Nous développons aussi un modèle in vitro 4D tumeur-sur-puce microfluidique en collaboration avec le Dr V. Senez (UMI2820 CNRS/LIMMS de l’institut ONCOLille). Les marqueurs d’intérêt identifiés seront validés pour leur potentiel thérapeutique sur les modèles murins précliniques. Nous étudierons particulièrement les canaux ioniques (TRPM7, KCa3.1; collab. Pr H. Ouadid-Aidouch, UPJV, Amiens) et les miARNs (miR-210, miR-132-3p, miR-96-5p) associés à l’expression de MUC4. Nous étudierons aussi le rôle des CAFS (collab. Dr C. Bousquet, CRCT Toulouse, INCa PAIR Pancreas). Nous voulons aussi, par l’utilisation du dispositif 4D microfluidique, augmenter le débit de criblage des drogues sur organoïdes développés à partir de biopsies obtenues pas aspiration à l’aiguille fine (collab. Dr J. Branche, service des maladies de l’appareil digestif, CHU Lille).
1.2.C. MUC4 comme cible thérapeutique ?
Nous développons aussi un projet structure-fonction visant à cibler le complexe oncogénique MUC4-ErbB2 via les domaines EGF de MUC4 (Liberelle 2019, Liberelle 2020) et développer à long terme des petites molécules inhibitrices à visée thérapeutique en collaboration avec les chimistes (Pr Lebègue, Centre LilNCog, UMR1172 Inserm, Lille).
2- Plasticité épigénétique de la cellule tumorale (Responsables : Dr Audrey Vincent, Dr Bernadette Neve; Co-investigateurs: Dr Guillemette Huet, Pr Pascal Pigny, Dr Julie Leclerc, Dr Anne-Frédérique Dessein, Dr Florence Renaud, Pr Stéphanie Truant)
Les modifications épigénétiques aberrantes sont connues pour contribuer à la capacité des cellules cancéreuses à allumer/éteindre les marqueurs à caractère souche générant une population cellulaire hétérogène au sein de la tumeur (Vincent & Van Seuningen 2012). Nous voudrions maintenant mieux caractériser ces mécanismes pour (i) identifier des modulateurs épigénétiques « druggable », (ii) identifier les tumeurs pouvant bénéficier d’inhibiteurs de première génération visant les modificateurs épigénétiques, (iii) concevoir de nouvelles épidrogues plus spécifiques de la plasticité/souchitude du cancer, (iv) inverser la chimiorésistance et (v) empêcher la prédisposition ou la reprise du cancer (Vincent 2019).
Pour cela, nous travaillons sur deux modèles, bien connus au laboratoire, que sont les cancers coliques et pancréatiques présentant des propriétés de souchitude différents. L’adénocarcinome pancréatique (PDAC) qui développe une chimorésistance très rapidement et le cancer colorectal (CCR) via la dormance et la rechute tumorales.
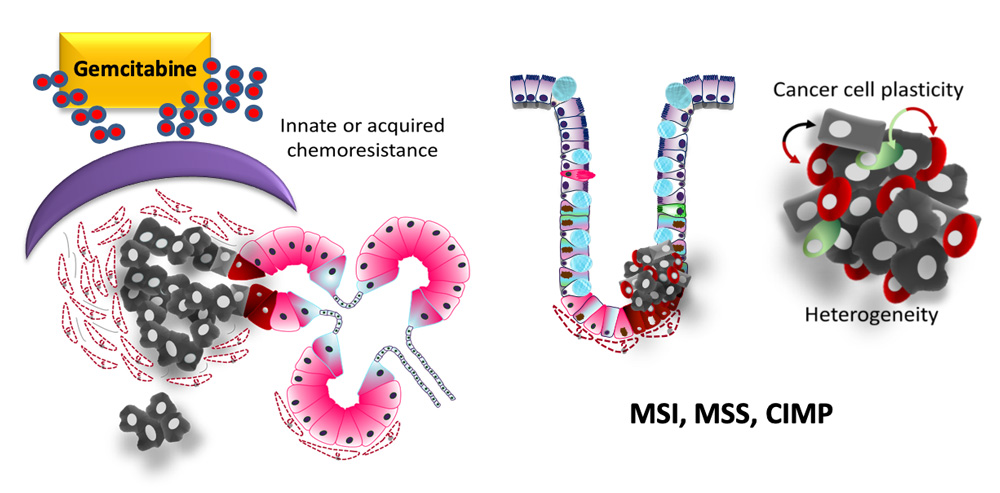 Le cancer du pancréas et chimiorésistance innée ou acquise (gauche). Plasticité de la cellule cancéreuse colique, dormance et hétérogénéité tumorales (droite)
Le cancer du pancréas et chimiorésistance innée ou acquise (gauche). Plasticité de la cellule cancéreuse colique, dormance et hétérogénéité tumorales (droite)
2.1. Modulateurs épigénétiques
Parmi les modulateurs épigénétiques directs, nous voulons identifier les ARN non codants (ARNnc) qui sont potentiellement responsables de la plasticité cellulaire (Neve 2018). Nous étudierons aussi l’impact du métabolisme sur les modifications de la chromatine par une approche de métabolomique (collab. Dr J.F. Goossens, plateforme spectrométrie de masse, U. Lille) et identifierons une signature métabolique de la plasticité cancéreuse (collab. Pr P. Marchetti/équipe leucémies Canther et plateau métabolisme de la cellule cancéreuse Canther, voir onglet services techniques). Nous évaluerons aussi le rôle du stress du réticulum (collab. Dr D. Delacour, UMR7592 CNRS, Institut Jacques Monod, Paris).
2.2. Modificateurs épigénétiques
Nous voulons établir les signatures des épienzymes et de la chromatine des cellules souches normales et cancéreuses par des approches à grande échelle (-omiques) dans les différents sous-types de CCR (CIN, MSI, CIMP) et de PDAC (épithélial vs de type basal). Nous utiliserons aussi des approches KD et d’édition du génome. Nous identifierons des interactions épienzyme-épienzyme potentiellement ciblables afin de découvrir de nouveaux composés chimiques spécifiques de l’état souche (collab. Pr A-S Voisin, CERMN, Univ. Caen, France).
2.3. Médiateurs épigénétiques
Nous allons continuer nos travaux sur les mécanismes et les conséquences de la méthylation des promoteurs de MLH1 et MUC5AC dans les formes familiales et sporadiques du CCR MSI (Leclerc 2018 ; Renaud 2015, 2016). Nous utiliserons des approches d’édition de l’épigénome (CRISPR-dCAs9), des vecteurs permettant de suivre la dynamique de méthylation de l’ADN à l’échelle de la cellule unique (plateau cellule unique ONCOLille) et par capture de la conformation de la chromatine (3C) (collab. plateformes génomique (Dr M. Figeac) & BiLille (Dr G. Marot), PLBS UMS2014-US41). Nous bénéficierons pour cela de la plus grande cohorte diagnostique française de patients atteints du syndrome de Lynch avec mutation du promoteur de MLH1 constitutionnelle (J. Leclerc, contrat d’interface Inserm 2020). A long terme, il s’agira de concevoir de nouvelles épidrogues spécifiques ciblant ces mécanismes (collab. Pr A-S Voisin, CERMN, Univ. Caen, France).
3- Profilage moléculaire de la récidive des cancers oeso-gastriques pour une meilleure prise en charge des patients (Responsable : Dr Florence Renaud; Co-investigateurs: Pr Marie-Pierre Buisine, Pr Guillaume Piessen, Dr Clarisse Eveno, Dr Isabelle Van Seuningen, Dr Deniz Pekin, Dr Fatima Lahdaoui)
Dans ce projet, nous voulons caractériser de manière précise le profil moléculaire de la tumeur primaire, de la récidive locale et de la métastase distante afin de comprendre (i) l’histoire naturelle des cancers oesogastriques (OG), (ii) les mécanismes de résistance aux traitements pour développer un test fonctionnel robuste utilisable en routine clinique, l’objectif final étant de proposer aux patients des traitements adaptés (médecine de précision).
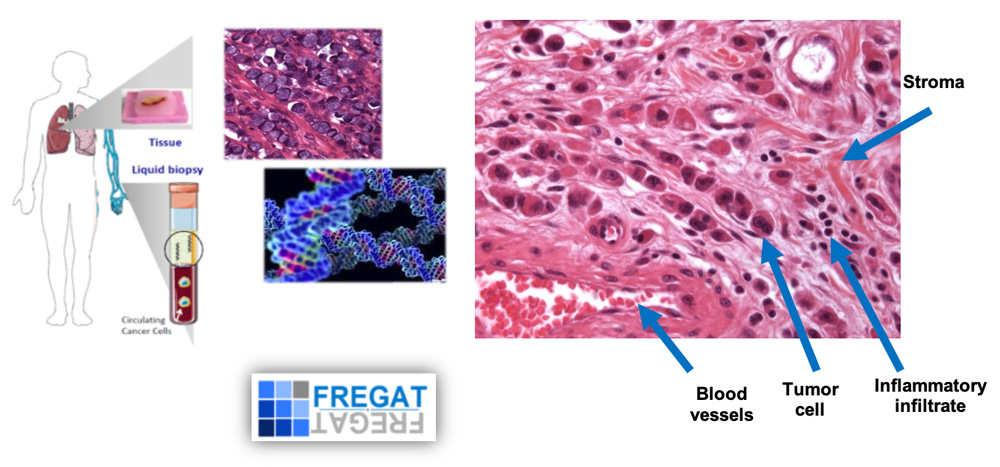 Profilage moléculaire de la tumeur primaire et de la récidive des cancers oesogastriques. Utilisation de la cohorte nationale BCB FREGAT (gauche). Hétérogénéités tumorale et cellulaire d’un carcinome gastrique à cellules indépendantes. Interactions tumeur-stroma (droite).
Profilage moléculaire de la tumeur primaire et de la récidive des cancers oesogastriques. Utilisation de la cohorte nationale BCB FREGAT (gauche). Hétérogénéités tumorale et cellulaire d’un carcinome gastrique à cellules indépendantes. Interactions tumeur-stroma (droite).
3.1. Recherche translationelle : Suivi moléculaire de la récidive pour anticiper la résistance de la maladie avancée
Par l’utilisation de nos données issues des études PROMOREC et REComics, nous voulons évaluer le suivi longitudinal l’ADN tumoral circulant (ADNtc) en utilisant des biopsies liquides non invasives déjà répertoriées dans la base de données clinico-biologique FREGAT, ceci comme substitut du profilage des récidives pour détecter de manière précoce la rechute et surmonter l’hétérogénéité intratumorale en routine clinique. Grâce au modèle Patient-Derived Organoids (PDO), nous évaluerons des drogues ciblant les marqueurs identifiés pour améliorer les traitements. Nous déterminerons aussi la signature UPR associée à la réponse à la chimiothérapie (collab. Dr O. Pluquet/équipe sénescence Canther).
3.2. Hétérogénéité génétique intratumorale dans le carcinome gastrique à cellules peu cohésives comme barrière à la médecine de précision
Une des raisons majeures d’échec des thérapies dans les cancers OG métastatiques est l’hétérogénéité intratumorale (HIT). Le carcinome gastrique à cellules peu cohésives est un sous type particulièrement agressif caractérisé par une incidence croissante et une forte chimiorésistance et récidive (Piessen 2013, 2009 ; Messager 2011). Nous initierons une étude pilote avec un large échantillonnage (cartographie) grâce à FREGAT afin d’établir la signature moléculaire aux niveaux ARN, ADN (collab. Pr M.P. Buisine, plateforme de génomique CHU de Lille & Dr M. Figeac, UMS2014-US41/PLBS) et protéique (collab. Pr I. Fournier, laboratoire PRISM, Villeneuve d’Ascq). L’HIT sera aussi définie par profilage moléculaire à partir de biopsies liquides (ADN) et nous évaluerons aussi la réaction stromale avant et après chimiothérapie. Enfin, nous évaluerons le profil immunitaire de la tumeur en réponse à l’oxaliplatine, un inducteur de la réponse immunitaire (collab. Dr N. Bonnefoy (IRCM, Inserm U1194, Montpellier) et Pr A. Adenis (service oncologie médicale, IRCM, Montpellier)).
3.3. Développement d’un dispositif 4D de type tumeur-sur-puce pour étudier la résistance aux drogues dans le cancer gastrique à cellules peu cohésives
Un modèle 4D microfluidque de tumeur-sur-puce sera réalisé (collaboration avec les physiciens UMI2820 CNRS/LIMMS de l’institut ONCOLille) (Pekin 2011) afin de mieux comprendre les mécanismes de résistance et pour développer un nouveau test fonctionnel pour une utilisation en routine clinique (résistance aux drogues, orientation du traitement).
3.4. Identifier les déterminants de la résistance tumorale et leurs conséquences sur la survie des patients
Les déterminants de la résistance de la tumeur, de la progression tumorale et des conséquences pour la survie du patient (réduire les symptômes affectifs et biologiques aux stades précoces de la maladie) sont des problèmes majeurs de santé publique (Lelorain 2018). Grâce à la BCB FREGAT, qui collecte aussi des données psychologiques des patients, nous pourrons évaluer l’association entre symptômes dépressifs et/ou d’anxiété avec (i) la morbimortalité post-opératoire et (ii) les marqueurs inflammatoires, immunitaires et tumoraux (collab. Pr V. Christophe, Dr D. Grynberg, SCALab, institut ONCOLille).
En conclusion, notre projet d’équipe devrait apporter de nouvelles connaissances fondamentales sur le rôle des mucines dans le cancer mais aussi sur les mécanismes contrôlant la résistance de la tumeur aux traitements chimiothérapeutiques, leur échappement à l’action des drogues, leur capacité à métastaser, le rôle du stroma et le rôle des modifications épigénétiques et des ARN non codants sur la plasticité de la cellule cancéreuse et sa capacité à se reprogrammer. Les projets interdisciplinaires avec les chimistes, les physiciens et les bioinformaticiens nous permettront de développer de nouveaux modèles originaux pour répondre à la question centrale de la résistance aux drogues. Les programmes translationnels avec les cliniciens, pathologistes et biologistes de l’équipe, par l’accès et l’utilisation de cohortes locales de patients annotées et de BCB nationales labellisées, et la collaboration avec les chercheurs en sciences humaines et sociales de l’institut ONCOLille devraient nous permettre de mieux définir les sous-types moléculaires des cancers que nous étudions et de trouver de nouveaux biomarqueurs pronostiques/prédictifs/de réponse afin de proposer une meilleure prise en charge, un meilleur suivi et des soins adaptés pour chaque patient.
> ARTICLES ORIGINAUX DU LABORATOIRE
> REVUES GÉNÉRALES
> ÉDITORIAL
> LETTRES – COMMENTAIRES
> CHAPITRES DE LIVRES
> ARTICLES ORIGINAUX ET REVUES GÉNÉRALES ISSUS DES COLLABORATIONS
> PUBLICATIONS CLINIQUES
ARTICLES ORIGINAUX DU LABORATOIRE
2024
Bourdon E*, Swierczewski T*, Goujon M, Boukrout N, Fellah S, Van der Hauwaert C, Larrue R, Lefebvre B, Van Seuningen I, Cauffiez C, Pottier N, Perrais M. MUC1 drives progression and chemoresistance of clear cell renal carcinomas, Cancers, 10.3390/cancers16020391
2022
Goujon M*, Woszczyk J*, Gaudelot K, Swierczewski T, Fellah S, Gibier JB, Van Seuningen I, Larrue R, Cauffiez C, Gnemmi V, Aubert S, Pottier N, Perrais M. A double negative feedback interaction between miR-21 and PPAR-a in clear renal cell carcinoma. Cancers, (Basel). 2022 Feb 4;14(3):795. doi: 10.3390/cancers14030795 IF: 6.639.
Hadj Bachir E*, Poiraud C*, Paget S, Stoup N, El Moghrabi S, Duchêne B, Jouy N, Bongiovanni A, Tardivel M, Weiswald LB, Vandepeutte M, Beugniez C, Escande F, Leteurtre E, OrgaRES consortium§, Poulain L, Lagadec C, Pigny P, Jonckheere N, Renaud F, Truant S, Van Seuningen I*, Vincent A*. A new pancreatic adenocarcinoma-derived organoid model of acquired chemoresistance to FOLFIRINOX: first insight of the underlying mechanisms. Biol Cell, 2022 Jan;114(1):32-55. doi: 10.1111/boc.202100003. IF: 4.458.
2021
Boukrout N, Souidi M, Lahdaoui F, Duchêne B, Neve B, Coppin L, Leteurtre E, Torrisani J, Van Seuningen I, Jonckheere N. Antagonistic roles of the tumor suppressor miR-210-3p and oncomucin MUC4 forming a negative feedback loop in pancreatic adenocarcinoma. Cancers (Basel), 2021 Dec 9;13(24):6197. doi: 10.3390/cancers13246197
Stoup N*, Liberelle M*, Schulz C, Cavdarli S, Vasseur R, Magnez R, Lahdaoui F, Skrypek, N, Peretti, F, Frénois F, Thuru X, Melnyk P, Renault N, Jonckheere N, Lebègue N, Van Seuningen I. The EGF Domains of MUC4 Oncomucin Mediate HER2 Binding Affinity and Promote Pancreatic Cancer Cell Tumorigenesis. Cancers (Basel), 2021 Nov 16;13(22):5746. doi: 10.3390/cancers13225746. IF: 6.639.
Gibier JB*, Swierczewski T*, Csanyi M, Hémon B, Glowacki FX, Maboudou P, Van Seuningen I, Cauffiez C, Pottier N, Aubert N, Perrais M, Gnemmi V. MUC1 mitigates renal injury and inflammation in endotoxin induced acute kidney injury by inhibiting the TLR4-MD2 axis and reducing pro-inflammatory macrophages infiltration. Shock, 2021 Oct 1;56(4):629-638. doi: 10.1097/SHK.0000000000001742.
Bauzone* M, Souidi M*, Dessein AF, Wisztorski M, Vincent A, Gimeno JP, Monté D, Van Seuningen I, Gespach C, Huet G. Cross-talk between YAP and RAR-RXR Drives Expression of Stemness Genes to Promote 5-FU Resistance and Self-Renewal in Colorectal Cancer Cells. Mol Cancer Res 2021 Apr;19(4):612-622. doi: 10.1158/1541-7786.MCR-20-0462.
2020
Jonckheere N, Auwercx J, Hadj Bachir E, Coppin L, Boukrout N, Vincent A, Neve B, Gautier M, Treviño V, Van Seuningen I. Unsupervised hierarchical clustering of pancreatic adenocarcinoma dataset from TCGA defines a mucin expression profile that impacts overall survival. Cancers (Basel). 2020 Nov 9;12(11):3309. doi: 10.3390/cancers12113309. IF: 6.126
Coppin L, Jannin A, Ait Yahya E, Thuillier C, Villenet C, Tardivel M, Bongiovanni A, Gaston C, de Beco S, Barois N, van Seuningen I, Durand E, Bonnefond A, Vienne JC, Vamecq J, Figeac M, Vincent A, Delacour D, Porchet N, Pigny P. Galectin-3 modulates epithelial cell adaptation to stress at the ER-mitochondria interface. Cell Death Dis. 2020 May 12;11(5):360. IF: 6.304
2019
Liberelle M, Magnez R, Thuru X, Bencheikh Y, Ravez S, Quenon C, Drucbert AS, Foulon C, Melnyk P, Van Seuningen I*, Lebègue N*. MUC4-ErbB2 Oncogenic Complex: Binding studies using Microscale Thermophoresis and Surface Plasmon Resonance. Sci Rep, 2019 Nov 13;9(1):16678. doi: 10.1038/s41598-019-53099-0
El Amrani M, Corfiotti F, Corvaisier M, Vasseur R, Fulbert M, Skrzypczyk C, Deshorgues AC, Gnemmi V, Tulasne D, Lahdaoui F, Vincent A, Pruvot FR, Van Seuningen I, Huet G, Truant S. Gemcitabine-induced epithelial-mesenchymal transition-like changes sustain chemoresistance of pancreatic cancer cells of mesenchymal-like phenotype. Mol Carcinog. 2019 Aug 2. doi: 10.1002/mc.23090.
2018
Jonckheere N, Van Seuningen I. Integrative analysis of the cancer genome atlas and cancer cell lines encyclopedia large-scale genomic databases: MUC4/MUC16/MUC20 signature is associated with poor survival in human carcinomas. J Transl Med. 2018 Sep 20;16(1):259. doi: 10.1186/s12967-018-1632-2.
Drubay V, Skrypek N, Cordiez L, Vasseur R, Schulz C, Boukrout N, Duchêne B, Coppin L, Van Seuningen I, Jonckheere N. TGF-βRII Knock-down in Pancreatic Cancer Cells Promotes Tumor Growth and Gemcitabine Resistance. Importance of STAT3 Phosphorylation on S727. Cancers (Basel). 2018 Jul 31;10(8). pii: E254. doi: 10.3390/cancers10080254.
2023
Jannin A, Dessein AF, Do Cao C, Vantyghem MC, Chevalier B, Van Seuningen I§, Jonckheere* N, Coppin* L. Metabolism of pancreatic neuroendocrine tumors: What can omics tell us? Front Endocrinol (Lausanne). 2023 Oct 16;14:1248575. doi: 10.3389/fendo.2023.1248575.
2021
Kumemura M, Pekin D, Menon VA, Van Seuningen I, Collard D, Tarhan MC. Fabricating silicon resonators for analysing biological samples. Micromachines, (Basel). 2021 Dec 12;12(12):1546. doi: 10.3390/mi12121546.
Jonckheere N, Neve B, Vincent A, Van Seuningen I. Mucin expression, epigenetic regulation and patient survival: A toolkit of prognostic biomarkers in epithelial cancers. Biochim Biophys Acta Rev Cancer. 2021 Aug;1876(1):188538. doi: 10.1016/j.bbcan.2021.188538.
Neve B, Jonckheere N, Vincent A, Van Seuningen I. Long non-coding RNAs: the tentacles of chromatin remodeler complexes. Cell Mol Life Sci, 2021 Feb;78(4):1139-1161. doi: 10.1007/s00018-020-03646-0.
Auwercx J, Rybarczyk P, Kischel P, Dhennin-Duthille I, Chatelain D, Sevestre H, van Seuningen I, Ouadid-Ahidouch H, Jonckheere N, Gautier M. Mg2+ transporters in digestive cancers. Nutrients. 2021 Jan 13;13(1):210. doi: 10.3390/nu13010210.
2020
Liberelle M, Jonckheere N, Melnyk P, Van Seuningen I, Lebegue N. EGF-containing Membrane-bound Mucins: a hidden ErbB2 targeting pathway? J Med Chem. 2020 May 28;63(10):5074-5088
2019
Vincent A, Ouelkdite-Oumouchal A, Souidi M, Leclerc J, Neve B, Van Seuningen I. Colon cancer stemness as a reversible epigenetic state: Implications for anticancer therapies. World J Stem Cells, 2019 Nov 26:11(11):920-936.
2018
Neve B, Jonckheere N, Vincent A, Van Seuningen I. Epigenetic Regulation by lncRNAs: An Overview Focused on UCA1 in Colorectal Cancer. Cancers (Basel). 2018 Nov 14;10(11). pii: E440. doi: 10.3390/cancers10110440.
Coppin L, Leclerc J, Vincent A, Porchet N, Pigny P. Messenger RNA life-cycle in cancer cells: Emerging role of conventional and non-conventional RNA-binding proteins? Int J Mol Sci, 2018, 19, 650; doi:10.3390/ijms19030650.
2019
Jonckheere N, Van Seuningen I. Fine-tuning autophagy in pancreatic adenocarcinoma: full blockage is required. Ann Transl Med. 2019 Mar;7(Suppl 1):S43.
2020
Neve B, Jonckheere N, Vincent A, Van Seuningen, I. 2020. Single cell analysis may shed new lights on the role of lncRNAs in chemo-resistance in gastrointestinal cancers. In: RNA technology. Springer Series. The Chemical Biology of Long Non Coding RNAs. Volume 10. Ed. S. Jurga & J. Barciszewski.
ARTICLES ORIGINAUX ET REVUES GÉNÉRALES ISSUS DES COLLABORATIONS
2024
2023
Quilbe A, Mustapha R, Duchêne B, Kumar A, Werkmeister E, Leteurtre E, Moralès O, Jonckheere N, Van Seuningen I, Delhem N. A novel anti-galectin-9 immunotherapy limits the early progression of pancreatic neoplastic lesions in transgenic mice. Front. Immunol. 14:1267279. doi: 10.3389/fimmu.2023.1267279
Cicero J, Trouvilliez S, Palma M, Ternier G, Decoster L, Happernegg H, Barois N, Van Outryve A, Dehouck L, Bourette RP, Adriaenssens E, Lagadec C, Tarhan CM, Collard D, Souguir Z, Vandenhaute E, Maubon G, Sipieter F, Borghi N, Shimizu F, Kanda T, Giacobini P, Gosselet F, Maubon N, Le Bourhis X, Van Seuningen I, Mysiorek C, Toillon RA. ProNGF promotes brain metastasis through TrkA/EphA2 induced Src activation in triple negative breast cancer cells. Exp Hematol Oncol. 2023 Dec 10;12(1):104. doi: 10.1186/s40164-023-00463-6.
2022
Dewaeles E, Carvalho K, Fellah S, Sim J, Boukrout N, Caillierez R, Ramakrishnan H, Van der Hauwaert C, Vijaya Shankara J, Martin N, Massri N, Launay A, Folger JK, de Schutter C, Larrue R, Loison I, Goujon M, Jung M, Le Gras S, Gomez-Murcia V, Faivre E, Lemaire J, Garat A, Beauval N, Maboudou P, Gnemmi V, Gibier JB, Buée L, Abbadie C, Glowacki F, Pottier N, Perrais M, Cunha RA, Annicotte JS, Laumet G, Blum D, Cauffiez C. Istradefylline protects from cisplatin-induced nephrotoxicity and peripheral neuropathy while preserving cisplatin antitumor effects. J Clin Invest. 2022 Nov 15;132(22):e152924. doi: 10.1172/JCI152924.
Larrue R, Fellah S, Van der Hauwaert C, Hennino MF, Perrais M, Lionet A, Glowacki F, Pottier N, Cauffiez C. The Versatile Role of miR-21 in Renal Homeostasis and Diseases. Cells. 2022 Nov 7;11(21):3525. doi: 10.3390/cells11213525
Auwercx J, Kischel P, Lefebvre T, Jonckheere N, Vanlaeys A, Guénin S, Radoslavova S, Van Seuningen I, Ouadid-Ahidouch H, Kocher H, Dhennin-Duthille I, Gautier M. TRPM7 Modulates Human Pancreatic Stellate Cell Activation. Cells 2022 Jul 21;11(14):2255. doi: 10.3390/cells11142255. IF : 7,666.
2021
Raby L, Völkel P, Hasanpour S, Cicero J, Toillon RA, Adriaenssens E, Van Seuningen I, Le Bourhis X, Angrand PO. Loss of Polycomb Repressive Complex 2 function alters digestive organ homeostasis and neuronal differentiation in zebrafish. Cells, 2021 Nov 12;10(11):3142. doi: 10.3390/cells10113142.
Wavelet-Vermuse C, Groux-Degroote S, Vicogne D, Cogez V, Venturi G, Trinchera M, Brysbaert G, Krzewinski-Recchi MA, Hadj Bachir E, Schulz C, Vincent A, Van Seuningen I, Harduin-Lepers A. Analysis of the proximal promoter of the human colon-specific B4GALNT2 (Sda synthase) gene: B4GALNT2 is transcriptionally regulated by ETS1. Biochim Biophys Acta Gene Regul Mech, 2021 Nov-Dec;1864(11-12):194747. doi: 10.1016/j.bbagrm.2021.194747.
2020
Roberti MP, Yonekura S, Duong CPM, Picard M, Ferrere G, Tidjani Alou M, Rauber C, Iebba V, Lehmann CHK, Amon L, Dudziak D, Derosa L, Routy B, Flament C, Richard C, Daillère R, Fluckiger A, Van Seuningen I, Chamaillard M, Vincent A, Kourula S, Opolon P, Ly P, Pizzato E, Becharef S, Paillet J, Klein C, Marliot F, Pietrantonio F, Benoist S, Scoazec JY, Dartigues P, Hollebecque A, Malka D, Pagès F, Galon J, Gomperts Boneca I, Lepage P, Ryffel B, Raoult D, Eggermont A, Vanden Berghe T, Ghiringhelli F, Vandenabeele P, Kroemer G, Zitvogel L. Chemotherapy-induced ileal crypt apoptosis and the ileal microbiome shape immunosurveillance and prognosis of proximal colon cancer. Nature Medicine, 2020 Jun;26(6):919-931. doi: 10.1038/s41591-020-0882-8. IF: 53.44
Lemaire J, Van der Hauwaert C, Savary G, Dewaeles E, Perrais M, Lo Guidice JM, Pottier N, Glowacki F, Cauffiez C. Cadmium-Induced Renal Cell Toxicity Is Associated With MicroRNA Deregulation. Int J Toxicol. 2020 Mar/Apr;39(2):103-114. doi: 10.1177/1091581819899039.
2019
Lambert M, Alioui M, Jambon S, Depauw S, Van Seuningen I, David-Cordonnier M-H. Direct and indirect targeting of HOXA9 transcription factor in acute myeloid leukemia. Cancers. 2019 Jun 17;11(6). IF: 5.326
2018
Vandenbussche C, Van der Hauwaert C, Dewaeles E, Franczak J, Hennino MF, Gnemmi V, Savary G, Tavernier Q, Nottet N, Paquet A, Perrais M, Blum D, Mari B, Pottier N, Glowacki F, Cauffiez C. Tacrolimus-induced nephrotoxicity in mice is associated with microRNA deregulation. Arch Toxicol. 2018 Jan 23. doi: 10.1007/s00204-018-2158-3
Aubert S, Berdelou A, Gnemmi V, Behal H, Caiazzo R, D’herbomez M, Pigny P, Wemeau JL, Carnaille B, Renaud F, Bouchindhomme B, Leteurtre E, Perrais M, Pattou F, Do Cao C. Large sporadic thyroid medullary carcinomas: predictive factors for lymph node involvement. Virchows Arch. 2018 Feb 1. doi: 10.1007/s00428-018-2303-7.
2022
2021
2020
Bitton L, Vandenbussche C, Wayolle N, Gibier JB, Cordonnier C, Verine J, Humez S, Bataille P, Lenain R, Ramdane N, Azar R, Mac Namara E, Hatron PY, Maurage CA, Perrais M, Frimat M, Vanhille P, Glowacki F, Buob D, Copin MC, Quéméneur T, Gnemmi V. Tubulointerstitial damage and interstitial immune cell phenotypes are useful predictors for renal survival and relapse in antineutrophil cytoplasmic antibody-associated vasculitis. J Nephrol. 2020 Aug;33(4):771-781. doi: 10.1007/s40620-019-00695-y. Epub 2020 Jan 8.
2019
Eveno C, Voron T, Piessen G. Laparoscopic Hyperthermic Intraperitoneal Chemotherapy is Safe for Patients with Peritoneal Metastases from Gastric Cancer and May Lead to Gastrectomy. Ann Surg Oncol. 2019 Jul 8. doi: 10.1245/s10434-019-07603-5.
Mariette C, Markar SR, Dabakuyo-Yonli TS, Meunier B, Pezet D, Collet D, D’Journo XB, Brigand C, Perniceni T, Carrère N, Mabrut JY, Msika S, Peschaud F, Prudhomme M, Bonnetain F, Piessen G; Fédération de Recherche en Chirurgie (FRENCH) and French Eso-Gastric Tumors (FREGAT) Working Group. Hybrid Minimally Invasive Esophagectomy for Esophageal Cancer. N Engl J Med. 2019 Jan 10;380(2):152-162. doi: 10.1056/NEJMoa1805101.
Renaud F, Chetboun M, Thevenet J, Delalleau N, Gmyr V, Hubert T, Bonner C, Messager M, Leteurtre E, Mariette C, Kerr-Conte J, Piessen G, Pattou F. Safety of Islet Autotransplantation After Pancreatectomy for Adenocarcinoma. Transplantation. 2019 Jan;103(1):177-181. doi: 10.1097/TP.0000000000002419.
2018
Mariette C, Renaud F, Piessen G, Gele P, Copin MC, Leteurtre E, Delaeter C, Dib M, Clisant S, Harter V, Bonnetain F, Duhamel A, Christophe V, Adenis A, on behalf of the FREGAT working group. The FREGAT biobank: a clinico-biological database dedicated to esophageal and gastric cancers. BMC Cancer 2018 Feb 6;18(1):139
Thèses en cours
- GOUJON Marine (D1 en 2021 – dir. thèse : Michaël PERRAIS)
- ROYER Felix (D1 en 2021 – dir. thèse : Vincent SENEZ – Isabelle VAN SEUNINGEN)
- MEYNARD Thomas (D1 en 2021 – dir. thèse : Isabelle VAN SEUNINGEN – Vincent SENEZ)
- HOUSSIER Robin (D1 en 2022 – dir. thèse : Vincent SENEZ)
- MOUM Leila (D1 en 2022 – dir. thèse : Nicolas LEBEGUE, Isabelle VAN SEUNINGEN)
- JANNIN Arnaud (D1 en 2021 – dir.thèse : Lucie COPPIN)
- HADJ-BACHIR Elsa (D2 en 2021 – dir. thèse : Audrey VINCENT)
- STOUP Nicolas (D3 en 2021 – dir. thèse : Isabelle VAN SEUNINGEN, Nicolas LEBEGUE)
- AUWERCX Julie (D1 en 2021 – dir.thèse : Mathieu GAUTIER, Nicolas JONCKHEERE)
Thèses soutenues
- STOUP Nicolas (23 février 2023) sous la co-direction d’Isabelle VAN SEUNINGEN et Nicolas LEBEGUE
- BOUKROUT Nihad (18 janvier 2022) sous la direction de Nicoals JONCKHEERE
- SOUIDI Mouloud (21 décembre 2021) sous la direction d’Isabelle VAN SEUNINGEN
- DE MIOLLIS Frédérick (28 janvier 2021) sous la co-direction d’Isabelle VAN SEUNINGEN et Vincent SENEZ
- SWIERCZEWSKI Thomas (26 janvier 2021) sous la direction de Michael PERRAIS
- GIBIER Jean-Baptiste (25 janvier 2021) sous la direction de Sébastien AUBERT et Viviane GNEMMI
- EL AMRANI Mehdi (11/12/2019) sous la direction de Isabelle VAN SEUNINGEN et Guillemette HUET
- SAAD Chadi (26/09/18) sous la direction de Marie-Pierre BUISINE et Hélène TOUZET
HDR soutenues
- GNEMMI Viviane (05/03/2020) sous la direction de Isabelle VAN SEUNINGEN