Équipe : Efficacité & Résistance aux Thérapies ciblées anti-tumorales
À LA UNE
Un champignon au secours des patients atteints de maladies génétiques rares.
Ces dix dernières années l’émergence des thérapies ciblées a profondément modifiée la prise en charge des patients présentant une addiction oncogénique, avec une amélioration de leur condition et de leur espérance de vie. Cependant, toutes les thérapies ciblées doivent faire face à l’émergence de résistances conduisant systématiquement à des échecs thérapeutiques. De plus, de nombreux cancers sont toujours diagnostiqués à un stade métastatique, une étape de la maladie ou les traitements sont peu efficaces et qui reste donc associée à un mauvais pronostic.
L’objectif global de notre équipe de recherche est de décrypter les mécanismes moléculaires conduisant à l’addiction oncogénique, puis au processus de résistance, dans le but d’améliorer l’efficacité des thérapies ciblées.
Nous concentrons notre attention sur le cancer du poumon et de la prostate et leur stade métastatique, représentant à eux deux la cause principale de mortalité par cancer. Nous tirons avantage de notre expertise sur des oncogènes dont l’implication dans chacun de ces cancers est bien documentée, incluant les récepteurs à activité tyrosine kinase (RTK) et les gènes de fusion ETS. Nous développons également de nouvelles approches thérapeutiques dans le but de corriger les mutations non-sens représentant de 5 à 40% des mutations affectants les gènes suppresseurs de tumeurs et responsable d’environ 10% des maladies génétiques.
Nous avons délibérément choisi de travailler en collaboration étroite avec les biologistes moléculaires, les pathologistes et les cliniciens du CHU de Lille qui sont directement impliqués dans la prise en charge des patients.
- Institut de Pathologie, CHU Lille, Pr MC Copin, Pr X Leroy, Dr S Humez
- Service d’Urologie, CHU Lille, Pr A Villers
- Service d’Oncologie Thoracique, CHU Lille, Pr A Cortot
- Service de Rhumathologie, Dr MH Vieillard
- Institut de Pathologie, Laboratoire d’Oncologie et de Génétique Moléculaire, CHU Lille, Pr F Escande, Dr C Descarpentries
- Plateau de génomique fonctionnelle et structurale, CHU Lille, Dr M Figeac
Nous accueillons régulièrement des étudiants en master et en thèse, des postdoctorants et des ingénieurs. Si vous souhaitez vous joindre à nous pour développer nos projets scientifiques, vous pouvez nous envoyer votre candidature.
> PERSONNEL CHERCHEURS
> PERSONNEL INGÉNIEURS/TECHNICIENS
> ÉTUDIANTS
 | David TULASNE – Directeur de recherche (DR2) INSERM |
 | Martine DUTERQUE-COQUILLAUD – Directeur de recherche (DR2) CNRS |
 | Anne CHOTTEAU-LELIEVRE – Professeure (PR2) Université de Lille |
 | Zoulika KHERROUCHE – Chercheur (CR1) Institut Pasteur de Lille |
 | Dr Marie José TRUONG, Chercheur Institut Pasteur de Lille |
 | Fabrice LEJEUNE – Chercheur (CRCN) INSERM |
 | Marie FERNANDES – Chercheur CDD-CNRS |
 | Marie-Hélène VIEILLARD – Onco-Rhumatologue (PH) COL and CHU Lille marie-helene.vieillard(@)chru-lille.fr |
 | Sarah HUMEZ – Anatomo-Pathologiste (PH) CHU Lille sarah.humez(@)chru-lille.fr |
 | Arnauld VILLERS – Urologue – Professeur (PU-PH) CHU Lille |
 | Xavier LEROY – Anatomo-Pathologiste – Professeur (PU-PH) CHU Lille |
 | Alexis CORTOT – Pneumologue– Professeur (PU-PH) CHU Lille |
 | Simon BALDACCI – Pneumologue (MCU-PH) – CHU Lille |
 | Anthony TURPIN – Oncologue CHU Lille |
PERSONNEL INGÉNIEURS/TECHNICIENS
 | Catherine AMPEN-GUFFROY – Ingénieure d’étude CNRS |
 | Gaëlle Rémy – Ingénieure CNRS Chargée de projets réglementaires des expérimentations in vivo. |
 | Anne-Claire FLOURENS – Technicienne de recherche INSERM |
 | Nathalie VANPOUILLE – Technicienne IPL |
 | Isabelle DAMOUR – Assistante de recherche IPL |
 | Audrey VINCHENT – Technicienne de recherche IPL |
 | Edwina GBAGUIDI-MICOURS – Assistante-Ingénieure IPL- CCD |
 | Clémence BURNICHON – Assistante-Ingénieure IPL- CCD |
 | Philippe JAMME – Étudiant en thèse Université de Lille Etude des mécanismes de résistance aux inhibiteurs tyrosine kinase dans les cancers pulmonaires porteurs d’une addiction oncogénique. Praticien hospitalo-universitaire en oncologie médicale au CHU de Lille, chef de clinique service de dermatologie, hopital Claude Hurriez. |
 | Martine PALMA – Étudiante en thèse Université de Lille |
 | Jonathan OLIVIER – Étudiant en thèse Université de Lille – Urologue CHU Lille jonathan.olivier(@)chru-lille.fr |
 | Aline GHESQUIER – Étudiante en thèse Université de Lille Cancer de la prostate et Covid-19 : rôle des androgènes dans la régulation de l’expression de TMPRSS2, protéase majeure dans l’infection virale au SARS-CoV-2 |
 | Célia GUERIN – Étudiante en thèse Université de Lille Etude fonctionnelle des nouvelles mutations du récepteur MET dans le cancer rénal héréditaire. |
 | Elisa CAROUGE – Étudiante en thèse Université de Lille |
Rémi TELLIER – Étudiant en thèse Université de Lille | |
 | Pauline PARENT – Étudiante en Master 2 Université de Lille |
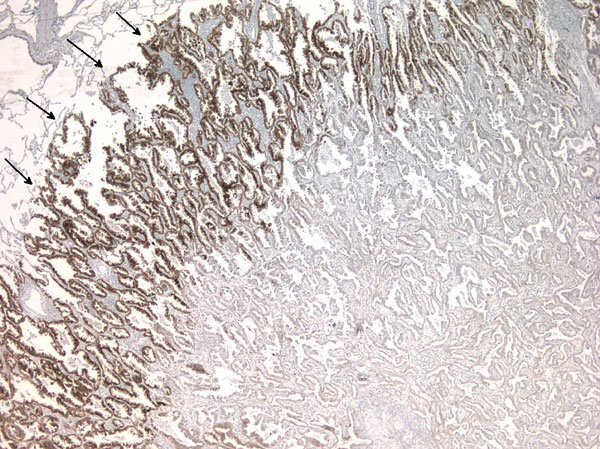
Dérégulation de MET dans le cancer du poumon
Tumeur pulmonaire exprimant le récepteur MET au front d’invasion
La dérégulation de MET est fréquemment observée dans les cancers et représente une cible majeure des nouvelles stratégies anti-tumorale. Les thérapies ciblées ont déjà démontré leur efficacité dans de nombreux cancers, comme par exemple les cancers de poumon non à petites cellules (NSCLC) présentant une mutation de l’EGFR. L’identification de nouvelles altérations moléculaires associées à un possible ciblage thérapeutique est toujours au cœur des préoccupations, comme le démontre la découverte récente des mutations affectants les sites d’épissage de l’exon 14 de MET conférant une sensibilité aux inhibiteur. Ainsi, nos objectifs principaux sont (i) comprendre les conséquences des régulations des RTK dans les cancers permettant une meilleur sélection des patients éligibles pour les thérapies ciblées, (ii) améliorer le diagnostic des altérations des RTK, (iii) anticiper les mécanismes de résistance pour proposer des stratégies thérapeutiques alternatives. Pour faire face à ces chalenges, nous développons une approche intégrée avec une collaboration forte avec les pathologistes, les plateformes de biologies moléculaire et les cliniciens du CHU de Lille.
Concernant plus spécifiquement les mutations des sites d’épissage de l’exon 14 de MET, représentant un nouveau type de mutations peu caractérisées, notre objectif est de comprendre précisément leurs conséquences fonctionnelles. Puisque les mutations MET exon 14 conduisent à la délétion du domaine juxtamembranaire, nous avons cherché à caractériser les mécanismes de régulations associées à ce domaine. Nous avons démontré que ce domaine subit plusieurs clivages protéolytiques affectant l’activité du récepteur. MET est notamment clivé par les caspases durant l’apoptose menant à la génération d’un fragment actif (p40MET) capable de favoriser la mort cellulaire via la perméabilisation mitochondriale (Lefebvre et al., Cell Death Dis, 2013). La relevance physiologique d’un tel clivage a été démontrée par le développement de souris Knock-in présentant une mutation de MET sur un des sites de clivage par les caspases. En effet, ces souris présentent une dérégulation de la balance survie/apoptose dans le foie (Duplaquet et al, eLife 2020). Nous avons également démontré que le domaine juxtamembranaire est ciblé par les gamma-sécrétases et les calpaïnes, des protéases impliquées dans la dégradation de MET par la génération de fragments labiles (Montagne et al., Oncotarget, 2017; Copin et al., Lung Cancer, 2016; Montagne et al., Cell Death Dis, 2015). Globalement, ces résultats démontrent que la perte du domaine juxtamembranaire de MET observée dans les cancers du poumon régule positivement l’activité de MET par l’inhibition de sa dégradation et de ses capacités pro-apoptotiques. Ces données renforcent le rationnel d’un ciblage de ces altérations dans le cancer du poumon. L’implication de MET dans la tumorigenèse incluant les mutation MET exon 14 est discuté dans les articles de synthèse suivant Duplaquet et al., Oncogene, 2018; Cortot et al., JNCI ,2017; Furlan et al., Cancer Res. 2014; Furlan et al., Hepatology, 2013.
Amélioration de la détection des altérations des RTK
Détection des mutations MET exon 14 chez des patients atteints d’un cancer du poumon non à petites cellules
La détection des mutations touchant les sites d’épissage de l’exon 14 de MET est rendue difficile par leur grande hétérogénéité et leur localisation majoritairement intronique. Cependant, par le biais de collaborations avec les plateformes de biologie moléculaire de Lille, les services de pathologie et d’Oncologie Thoracique, nous avons optimisé le design d’un panel de séquençage haut-débit permettant la détection de la majorité des altérations de MET. Ce panel utilisé maintenant en routine clinique a permis d’augmenter le taux de détection de 0.3% à 2.2% chez les patients atteints d’un NSCLC (Descarpentries et al., J Thorac Oncol, 2018, Baldacci et al., J Thorac Oncol, 2020). Les patients présentant ces mutations bénéficient maintenant des thérapies ciblées anti-MET notamment par le biais d’essais cliniques (VISION trial, Merck Serrono).
Dans le cadre d’une approche plus globale, nous avons étendu nos recherches des altérations à l’ensemble des membres de la famille des RTK. Nous nous sommes intéressés aux cancers colorectaux métastatiques dans lesquelles les mutations des RTK sont peu décrites. Des appariements précieux d’échantillons de tissus sain/tumeur/métastase ont été collectés dans les hôpitaux du nord de la France avec le soutien du Cancéropôle Nord-Ouest. Bien que le séquençage haut-débit ait révélé de multiples nouvelles mutations des RTK, une analyse fonctionnelle systématique n’a pas mis en évidence de mutations activatrices. Ces résultats soulignent l’importance des études fonctionnelles pour valider la présence de mutations activatrices ciblables (Duplaquet et al, Mol Cancer Ther, 2019).
Le récepteur MET dans la résistance aux traitements
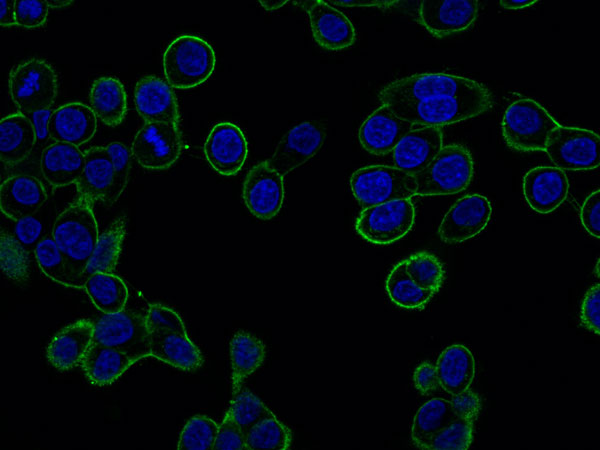
Cellules cancéreuses GTL16 exprimant le récepteur MET à la membrane plasmique
Le récepteur MET est un driver oncogénique dans de nombreux cancers, il est également impliqué dans la résistance aux inhibiteurs de tyrosine kinase (ITK) dirigés contre d’autres récepteurs. Ainsi, le traitement des cancers bronchiques non à petites cellules (CBNPC) mutés sur le récepteur EGFR repose sur les ITK-EGFR. Cependant tous les patients traités par ITK-EGFR finissent par présenter une progression tumorale, du fait de mécanismes de résistance.
L’amplification du gène MET est l’une des altérations les plus fréquentes dans la résistance acquise aux inhibiteurs d’EGFR des CBNPC.
Afin de comprendre l’impact de cette amplification, nous avons étudié les modifications phénotypiques induites par l’activation de MET dans des lignées cellulaires dérivées de carcinomes bronchiques et des modèles murins. Nous avons démontré que l’amplification de MET ne confère pas uniquement aux cellules sous traitement une signalisation alternative responsable de la résistance aux ITK-EGFR; l’amplification de MET confère également aux cellules tumorales un phénotype plus agressif et invasif qui favorise la métastase, notamment par l’activation des facteurs de transcription PEA3 de la famille ETS (Kherrouche et al., Mol Oncol. 2015; Baldacci, Kherrouche et al., Lung Cancer, 2018).
De plus, l’étude des données cliniques d’une cohorte nationale de patients porteurs d’un CBNPC EGFR muté métastatique, a révélé un délai d’apparition de nouvelles métastases plus court après progression sous traitement ITK-EGFR pour les patients porteurs d’une amplification de MET par rapport aux autres patients (Baldacci et al., Oncotarget, 2017). Nos résultats plaident en faveur d’une utilisation précoce d’inhibiteurs de MET en association avec les ITK-EGFR afin d’éviter l’émergence d’un clone tumoral résistant plus agressif.
Récemment, des mutations responsables de l’épissage de l’exon 14 du récepteur MET (METex14) ont été détectées dans les cancers bronchiques, colorectaux et gastriques. Ces mutations sont associées à une efficacité des inhibiteurs de MET, à un niveau toutefois moindre que dans d’autres indications d’ITK, suggérant l’existence de mécanismes de résistance primaire aux traitements. Grâce à des modèles cellulaires portant une mutation METex14 et une mutation activatrice de PIK3CA ou une perte d’expression de PTEN, nous avons montré que l’activation de la voie de signalisation PI3K constituait un mécanisme de résistance aux ITK-MET. De manière intéressante, l’inhibition de la voie PI3K permettait de restaurer la sensibilité aux ITK-MET. Nos travaux ouvrent la voie pour de nouvelles stratégies thérapeutiques visant à contrecarrer la résistance aux ITK-MET (Jamme et al, J Thorac Oncol, 2020).
Correction des mutations non-sens dans les cancers et autres maladies génétiques
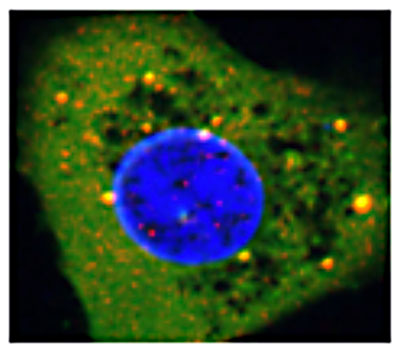
Colocalisation dans des foyers cytoplasmiques de cellules HeLa de UPF1 (vert) et de DCP1a (rouge) suite à un traitement par la colchicine
Les mutations non-sens changent un codon en un codon stop prématuré. La conséquence de la présence d’un codon stop prématuré est l’absence d’expression du gène correspondant du fait de l’activation d’un mécanisme de contrôle qualité appelé « nonsense-mediated mRNA decay (NMD) ». Environ 10% des patients souffrant d’une maladie génétique sont porteurs d’un codon stop prématuré à l’origine de leur pathologie. Parmi les maladies génétiques, on trouve les maladies rares comme la mucoviscidose, la myopathie de Duchenne, le syndrome de Rett, etc… ainsi que des pathologies plus fréquentes comme les désordres métaboliques et neurologiques et les cancers.
Au laboratoire, nous recherchons des molécules capables de restaurer l’expression de gènes porteurs d’une mutation non-sens. Pour cela, nous avons construit des systèmes de criblage pour identifier des molécules capables d’inhiber le NMD et/ou activer la translecture des codons stop prématurés (Gonzales et al., Orphanet Journal of Rare diseases 2012, Benhabiles et al., Plos One 2017, Trzaska et al., Nature Commun 2020). Les molécules sélectionnées par criblage sont ensuite testées sur des modèles cellulaires et murins porteurs d’une mutation non-sens dans le gène TP53, PTEN, CFTR ou dystrophine afin de caractériser ces molécules et comprendre leur mode d’action. Notre objectif est de développer de nouvelles approches thérapeutiques pour les maladies génétiques causées par des mutations non-sens et d’améliorer notre connaissance des mécanismes de reconnaissance des codons stop prématurés et de leur régulation. Nous avons ainsi montré que le NMD est inhibé durant l’apoptose du fait du clivage de deux facteurs principaux du NMD (UPF1 et UPF2) par des caspases (Jia et al., Cell Death and Diff, 2015).
Nous avons aussi montré que les perturbateurs du cytosquelette sont des inhibiteurs du NMD très efficaces. L’étude de ces molécules nous a conduit à identifier et caractériser de nouveaux foyers cytoplasmiques dans lesquels la translecture se déroule et que nous avons appelés foyers de translecture (Jia et al., J Cell Science, 2017).
Métastases osseuses et facteurs de transcription ETS dans le cancer de la prostate
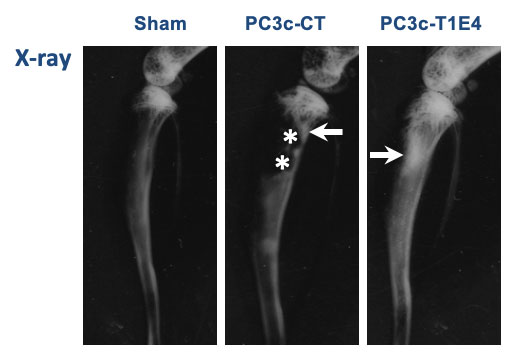
Métastases osseuses en modèle animal de lignées cellulaires prostatiques exprimant ou non la fusion ERG
Le cancer de la prostate est le plus fréquent chez l’homme dans les pays occidentaux. Une tumeur locale est associée à une évolution lente et une bonne survie alors que les stades plus avancés révèlent la présence de métastases, osseuses dans 80% des cas, phase incurable et dévastatrice et principale cause de mortalité des cancers de la prostate.
L’absence de biomarqueurs permettant de suivre la survenue des métastases osseuses est un frein majeur à l’amélioration des thérapies. Le cancer de la prostate dépend des androgènes. Pour exercer son action, l’hormone 5alpha-dihydrostestostérone (DHT) se fixe au récepteur aux androgènes (AR), membre de la famille des récepteurs nucléaires, qui joue un rôle-clé dans la prolifération et la survie des cellules cancéreuses. Les patients atteints de stades avancés reçoivent une hormonothérapie qui consiste à une privation androgénique par castration médicale. Une phase de résistance à la castration est alors inéluctable et apparaît après 2 à 3 ans. Plusieurs mécanismes de résistance à la castration ont déjà été décrits dans la littérature et entraînent une activation constitutionnelle du récepteur aux androgènes en l’absence ou en présence d’une faible quantité de DHT. A ce stade de la maladie, l’évolution s’accélère et les options thérapeutiques sont limitées.
En 2005, la découverte de gènes de fusion, issus de remaniements chromosomiques, a ouvert une nouvelle voie dans la compréhension du processus de cancérisation de la prostate. Ces fusions impliquent d’une part la partie promotrice non-codante du gène hormono-dépendant TMPRSS2 (Transmembrane protease serine2), et d’autre part la partie codante des gènes de la famille des facteurs de transcription ETS, le gène ERG (ETS Related Gene) dans plus de 50% des cas, et les gènes ETV dans environ 10% des cas. L’expression de ces facteurs de transcription constituent un réseau anormal de régulation qui conduit au déséquilibre du transcriptome des cellules tumorales.
Notre équipe a montré que l’expression des fusions ERG augmente la migration et l’invasion des cellules tumorales métastatiques, perturbant le transcriptome des cellules tumorales, en particulier la MMP9 et l’Endothéline, connue pour être impliquée dans la formation des lésions ostéoblastiques (Tian et al., Oncogene, 2014). Des expériences in vivo, d’induction de lésions osseuses par injections intratibiales ou intracardiaques, ont montré que l’expression de la fusion est capable d’augmenter respectivement le caractère osteoblastique de lésions et le nombre des métastases localisées au niveau du squelette (Deplus et al., Oncotarget, 2017 ; Delliaux, Tian, et al., Cancer letters, 2018). A partir de ces résultats, nous avons montré que la fusion ERG augmente, en régulant directement, des marqueurs ostéoblastiques majeurs, le Collagène de type1 et la Phosphatase alcaline.
Ces résultats ont été confortés chez l’homme grâce à notre étroite collaboration avec les cliniciens, oncologues, anatomo-pathologistes, urologues et onco-rhumatologues du CHU et Centre Oscar Lambret de Lille.
En parallèle, nous avons étudié le rôle des facteurs ETV dans la tumorigenèse et les métastases du cancer du sein, un autre cancer hormono-dépendant associé fréquemment aux métastases osseuses. Nous avons montré que, parmi les mécanismes moléculaires contrôlés par le facteur de transcription ETV4, le gène MMP13, codant une métalloprotéase extracellulaire, est un gène cible direct. ETV4 participe à la formation tumorale induite par ETV4 in vivo en perturbant la prolifération, la migration et l’invasion cellulaires. Enfin, la co-expression de ETV4 et MMP13 est associée à un mauvais pronostic du cancer du sein (Dumortier et al., Breast Cancer Res., 2018). De manière intéressante, MMP13 a été montré jouer un rôle direct dans la digestion de la matrice extracellulaire en complément de l’activité de MMP9 et d’autres enzymes. MMP13 est également impliqué dans la formation des lésions ostéolytiques du cancer du sein.
L’ensemble de ces résultats révèle un rôle encore inconnu des facteur ETS, en particulier des fusions ERG, dans le tropisme osseux et le caractère ostéoblastique dans la formation des métastases osseuses du cancer de prostate.
Notre objectif est maintenant de définir une signature d’expression génique, outil de diagnostic et de suivi de la progression tumorale et de l’évolution métastatique, afin d’améliorer le choix des thérapies les plus adaptées à chaque patient atteint du cancer de la prostate.
2023
Dehem A, Mazieres J, Chour A, Guisier F, Ferreira M, Boussageon M, Girard N, Moro-Sibilot D, Cadranel J, Zalcman G, Ricordel C, Wislez M, Munck C, Poulet C, Gauvain C, Descarpentries C, Wasielewski E, Cortot AB, Baldacci S. Characterization of 164 patients with NRAS mutated non-small cell lung cancer (NSCLC). Lung Cancer. 2023 Oct 9;186:107393. doi: 10.1016/j.lungcan.2023.107393. Online ahead of print.PMID: 37839252
Chour A, Denis J, Mascaux C, Zysman M, Bigay-Game L, Swalduz A, Gounant V, Cortot A, Darrason M, Fallet V, Auclin E, Basse C, Tissot C, Decroisette C, Bombaron P, Giroux-Leprieur E, Odier L, Brosseau S, Creusot Q, Gueçamburu M, Meersseman C, Rochand A, Costantini A, Gaillard CM, Wasielewski E, Girard N, Cadranel J, Lafitte C, Lebossé F, Duruisseaux M. Brief Report: Severe Sotorasib-Related Hepatotoxicity and Non-Liver Adverse Events Associated With Sequential Anti-Programmed Cell Death (Ligand)1 and Sotorasib Therapy in KRASG12C-Mutant Lung Cancer. J Thorac Oncol. 2023 Oct;18(10):1408-1415. doi: 10.1016/j.jtho.2023.05.013. Epub 2023 May 20. PMID: 37217096.
Khiter F, Kherrouche Z, Dubois V, Slupek S, Petit E, Debrie AS, Cauchi S, Barois N, Rouanet C, Mielcarek N. Combined regulation of pro-inflammatory cytokines production by STAT3 and STAT5 in a model of B. pertussis infection of alveolar macrophages. Front Immunol. 2023 Sep 28;14:1254276. doi: 10.3389/fimmu.2023.1254276. eCollection 2023.
Ardin C, Humez S, Leroy V, Ampere A, Bordier S, Escande F, Turlotte A, Stoven L, Nunes D, Cortot A, Gauvain C. Pursuit or discontinuation of anti-PD1 after 2 years of treatment in long-term responder patients with non-small cell lung cancer. Ther Adv Med Oncol. 2023 Sep 13;15:17588359231195600. doi: 10.1177/17588359231195600.
Mazieres J, Paik PK, Garassino MC, Le X, Sakai H, Veillon R, Smit EF, Cortot AB, Raskin J, Viteri S, Wu YL, Yang JCH, Ahn MJ, Ma R, Zhao J, O’Brate A, Berghoff K, Bruns R, Otto G, Johne A, Felip E, Thomas M. Tepotinib Treatment in Patients With MET Exon 14-Skipping Non-Small Cell Lung Cancer: Long-term Follow-up of the VISION Phase 2 Nonrandomized Clinical Trial. JAMA Oncol. 2023 Sep 1;9(9):1260-1266. doi: 10.1001/jamaoncol.2023.1962. PMID: 37270698.
Fernandes M, Paget S, Kherrouche Z, Truong MJ, Vinchent A, Meneboo JP, Sebda S, Werkmeister E, Descarpentries C, Figeac M, Cortot AB and Tulasne. D. Transforming properties of MET receptor exon 14 skipping can be recapitulated by loss of the CBL ubiquitin ligase binding site. FEBS Lett. 2023 Jul 19. doi: 10.1002/1873-3468.14702.
Delval L, Hantute-Ghesquier A, Sencio V, Flaman JM, Robil C, Angulo FS, Lipskaia L, Çobanoğlu O, Lacoste AS, Machelart A, Danneels A, Corbin M, Deruyter L, Heumel S, Idziorek T, Séron K, Sauve F, Bongiovanni A, Prévot V, Wolowczuk I, Belouzard S, Saliou JM, Gosset P, Bernard D, Rouillé Y, Adnot S, Duterque-Coquillaud M, Trottein F. Removal of senescent cells reduces the viral load and attenuates pulmonary and systemic inflammation in SARS-CoV-2-infected, aged hamsters. Nat Aging. 2023 Jul;3(7):829-845. doi: 10.1038/s43587-023-00442-w.
Tsuboi M, Herbst RS, John T, Kato T, Majem M, Grohé C, Wang J, Goldman JW, Lu S, Su WC, de Marinis F, Shepherd FA, Lee KH, Le NT, Dechaphunkul A, Kowalski D, Poole L, Bolanos A, Rukazenkov Y, Wu YL; ADAURA Investigators. Overall Survival with Osimertinib in Resected EGFR-Mutated NSCLC. N Engl J Med. 2023 Jul 13;389(2):137-147. doi: 10.1056/NEJMoa2304594. Epub 2023 Jun 4.PMID: 37272535.
L Brunet, D Alexandre, J Lee, MM Blanquer-Rossello, A Guernet, H. Chhouri, Z. Kherrouche, A. Arabo, S. Yao, D. Godefroy, J. Dehedin, J. Li, C. Duparc, P. Jamme, A. Vinchent, C. Berard, D. Tulasne, S. Arena, A. Bardelli, C. Cheng, B. Chul Cho, C. Coulouarn, S. Aaronson, AB. Cortot, Y. Anouar, L. Grumolato. Prolonging lung cancer response to EGFR inhibition by targeting the selective advantage of resistant cells. bioRxiv, 2023.06. 19.545595
Baudry AS, Charton E, Piessen G, Vanlemmens L, Cortot A, Ceban T, Anota A, Christophe V. Emotional distress, supportive care needs and age in the prediction of quality of life of cancer patients’ caregivers: A cross-sectional study. Eur J Oncol Nurs. 2023 Jun;64:102324. doi: 10.1016/j.ejon.2023.102324.
Fernandes M, Hoggard B, Jamme P, Paget S, Truong MJ, Grégoire V, Vinchent A, Descarpentries C, Morabito A, Stanislovas J, Farage E, Meneboo JP, Sebda S, Bouchekioua-Bouzaghou K, Nollet M, Humez S, Perera T, Fromme P, Grumolato L, Figeac M, Copin MC, Tulasne D, Cortot AB, Kermorgant S, Kherrouche Z. MET exon 14 skipping mutation is a hepatocyte growth factor (HGF)-dependent oncogenic driver in vitro and in humanised HGF knock-in mice. Mol Oncol. 2023 Feb 17. doi: 10.1002/1878-0261.13397.
Bogard G, Barthelemy J, Hantute-Ghesquier A, Sencio V, Brito-Rodrigues P, Séron K, Robil C, Flourens A, Pinet F, Eberlé D, Trottein F, Duterque-Coquillaud M, Wolowczuk I. SARS-CoV-2 infection induces persistent adipose tissue damage in aged golden Syrian hamsters. Cell Death Dis. 2023 Feb 1;14(2):75. doi: 10.1038/s41419-023-05574-w.
2022
Mushtaq R, Cortot AB, Gautschi O, Mazieres J, Camidge DR. PD-1/PD-L1 inhibitor activity in patients with gene-rearrangement positive non-small cell lung cancer-an IMMUNOTARGET case series. Transl Lung Cancer Res. 2022 Dec;11(12):2412-2417. doi: 10.21037/tlcr-22-329.
Christophe V, Anota A, Vanlemmens L, Cortot AB, Ceban T, Piessen G, Charton E, Baudry AS. Unmet supportive care needs of caregivers according to medical settings of cancer patients: a cross-sectional study. Support Care Cancer. 2022 Nov;30(11):9411-9419. doi: 10.1007/s00520-022-07379-7.
Debieuvre D, Molinier O, Falchero L, Locher C, Templement-Grangerat D, Meyer N, Morel H, Duval Y, Asselain B, Letierce A, Trédaniel J, Auliac JB, Bylicki O, Moreau L, Fore M, Corre R, Couraud S, Cortot AB; Study Group KBP-2020-CPHG; KBP-2020-CPHG. Lung cancer trends and tumor characteristic changes over 20 years (2000-2020): Results of three French consecutive nationwide prospective cohorts’ studies. Lancet Reg Health Eur. 2022 Aug 29;22:100492. doi: 10.1016/j.lanepe.2022.100492.
Veillon R, Sakai H, Le X, Felip E, Cortot AB, Smit EF, Park K, Griesinger F, Britschgi C, Wu YL, Melosky B, Baijal S, Jr GC, Sedova M, Berghoff K, Otto G, Paik PK. Safety of Tepotinib in Patients With MET Exon 14 Skipping NSCLC and Recommendations for Management. Clin Lung Cancer. 2022 Jun;23(4):320-332. doi: 10.1016/j.cllc.2022.03.002.
Baldacci S, Besse B, Avrillon V, Mennecier B, Mazieres J, Dubray-Longeras P, Cortot AB, Descourt R, Doubre H, Quantin X, Duruisseaux M, Monnet I, Moro-Sibilot D, Cadranel J, Clément-Duchêne C, Cousin S, Ricordel C, Merle P, Otto J, Schneider S, Langlais A, Morin F, Westeel V, Girard N. Lorlatinib for advanced anaplastic lymphoma kinase-positive non-small cell lung cancer: Results of the IFCT-1803 LORLATU cohort. Eur J Cancer. 2022 May;166:51-59. doi: 10.1016/j.ejca.2022.01.018.
Cortot AB, Le X, Smit E, Viteri S, Kato T, Sakai H, Park K, Camidge DR, Berghoff K, Vlassak S, Paik PK. Safety of MET Tyrosine Kinase Inhibitors in Patients With MET Exon 14 Skipping Non-small Cell Lung Cancer: A Clinical Review. Clin Lung Cancer. 2022 May;23(3):195-207. doi: 10.1016/j.cllc.2022.01.003.
Sebai M, Tulasne D, Caputo SM, Verkarre V, Fernandes M, Guérin C, Reinhart F, Adams S, Maugard C, Caron O, Guillaud-Bataille M, Berthet P, Bignon YJ, Bressac-de Paillerets B, Burnichon N, Chiesa J, Giraud S, Lejeune S, Limacher JM, de Pauw A, Stoppa-Lyonnet D, Zattara-Cannoni H, Deveaux S, Lidereau R, Richard S, Rouleau E. Novel germline MET pathogenic variants in French patients with papillary renal cell carcinomas type I. Hum Mutat. 2022 Mar;43(3):316-327. doi: 10.1002/humu.24313.
Sencio V, Machelart A, Robil C, Benech N, Hoffmann E, Galbert C, Deryuter L, Heumel S, Hantute-Ghesquier A, Flourens A, Brodin P, Infanti F, Richard V, Dubuisson J, Grangette C, Sulpice T, Wolowczuk I, Pinet F, Prévot V, Belouzard S, Briand F, Duterque-Coquillaud M, Sokol H, Trottein F. Alteration of the gut microbiota following SARS-CoV-2 infection correlates with disease severity in hamsters. Gut Microbes. 2022 Jan-Dec;14(1):2018900. doi: 10.1080/19490976.2021.2018900.
Palma M, Leroy C, Salomé-Desnoulez S, Werkmeister E, Kong R, Mongy M, Le Hir H, Lejeune F. A role for AKT1 in nonsense-mediated mRNA decay. Nucleic Acids Res. 2021 Nov 8;49(19):11022-11037. doi: 10.1093/nar/gkab882.
Le X, Sakai H, Felip E, Veillon R, Garassino MC, Raskin J, Cortot AB, Viteri S, Mazieres J, Smit EF, Thomas M, Iams WT, Cho BC, Kim HR, Yang JC, Chen YM, Patel JD, Bestvina CM, Park K, Griesinger F, Johnson M, Gottfried M, Britschgi C, Heymach J, Sikoglu E, Berghoff K, Schumacher KM, Bruns R, Otto G, Paik PK. Tepotinib Efficacy and Safety in Patients with MET Exon 14 Skipping NSCLC: Outcomes in Patient Subgroups from the VISION Study with Relevance for Clinical Practice. Clin Cancer Res. 2021 Nov 17. doi: 10.1158/1078-0432.CCR-21-2733.
Vary A, Lebellec L, Di Fiore F, Penel N, Cheymol C, Rad E, El Hajbi F, Lièvre A, Edeline J, Bimbai AM, Le Deley MC, Turpin A. FOLFIRINOX relative dose intensity and disease control in advanced pancreatic adenocarcinoma. Ther Adv Med Oncol. 2021 Jul 16;13:17588359211029825. doi: 10.1177/17588359211029825.
Pinquie F, Cortot AB, Chevalier LM, Morel A, Sandrini J, Guguen C, Morvan B, Molinier O. A Case Report of Successful Treatment With Crizotinib to Overcome Resistance to Osimertinib in an EGFR Mutated Non-Small-Cell Lung Cancer Patient Harboring an Acquired MET Exon 14 Mutation. Clin Lung Cancer. 2021 Jun 13:S1525-7304(21)00142-X. doi: 10.1016/j.cllc.2021.06.002.
Cortot AB, Madroszyk A, Giroux-Leprieur E, Molinier O, Quoix E, Bérard H, Otto J, Rault I, Moro-Sibilot D, Raimbourg J, Amour E, Morin F, Hureaux J, Moreau L, Debieuvre D, Morel H, Renault A, Pichon E, Huret B, Charpentier S, Denis MG, Cadranel J. First-Line Afatinib plus Cetuximab for EGFR-Mutant Non-Small Cell Lung Cancer: Results from the Randomized Phase II IFCT-1503 ACE-Lung Study. Clin Cancer Res. 2021 Aug 1;27(15):4168-4176. doi: 10.1158/1078-0432.CCR-20-4604.
Fernandes M, Jamme P, Cortot AB, Kherrouche Z, Tulasne D. When the MET receptor kicks in to resist targeted therapies. Oncogene. 2021 Jun;40(24):4061-4078. doi: 10.1038/s41388-021-01835-0.
Jeschke J, Collignon E, Al Wardi C, Krayem M, Bizet M, Jia Y, Garaud S, Wimana Z, Calonne E, Hassabi B, Morandini R, Deplus R, Putmans P, Dube G, Singh NK, Koch A, Shostak K, Rizzotto L, Ross RL, Desmedt C, Bareche Y, Rothé F, Lehmann-Che J, Duterque-Coquillaud M, Leroy X, Menschaert G, Teixeira L, Guo M, Limbach PA, Close P, Chariot A, Leucci E, Ghanem G, Yuan BF, Willard-Gallo K, Sotiriou C, Marine JC, Fuks F. Downregulation of the FTO m6A RNA demethylase promotes EMT-mediated progression of epithelial tumors and sensitivity to Wnt inhibitors. Nat Cancer. 2021 Jun;2(6):611-628. doi: 10.1038/s43018-021-00223-7.
Vauléon E, Behal H, Lebellec L, Desbarbieux R, Baldacci S, Simon N, Pannier D, Vieillard MH, Turpin A. Does bevacizumab increase joint pain in patients with cancer? Results of the prospective observational BEVARTHRALGIA study. Cancer Chemother Pharmacol. 2021 Apr;87(4):533-541. doi: 10.1007/s00280-020-04226-6.
Palma M, Lejeune F. Deciphering the molecular mechanism of stop codon readthrough. Biol Rev Camb Philos Soc. 2021 Feb;96(1):310-329. doi: 10.1111/brv.12657. Epub 2020 Oct 22. PMID: 33089614.
2020
Paik PK, Felip E, Veillon R, Sakai H, Cortot AB, Garassino MC, Mazieres J, Viteri S, Senellart H, Van Meerbeeck J, Raskin J, Reinmuth N, Conte P, Kowalski D, Cho BC, Patel JD, Horn L, Griesinger F, Han JY, Kim YC, Chang GC, Tsai CL, Yang JC, Chen YM, Smit EF, van der Wekken AJ, Kato T, Juraeva D, Stroh C, Bruns R, Straub J, Johne A, Scheele J, Heymach JV, Le X. Tepotinib in Non-Small-Cell Lung Cancer with MET Exon 14 Skipping Mutations. N Engl J Med. 2020 May 29. doi: 10.1056/NEJMoa2004407. IF 70.670
Bouchet M, Lainé A, Boyault C, Proponnet-guerault M, Meugnier E, Bouazza l, Kan C, Geraci S, El moghrabi S, Hernandez-vargas MH, Benetollo C, Yoshiko Y, Duterque-coquillaud M, Clézardin P, Marie JC and Bonnelye E. ERRα Expression in Bone Metastases Leads to an Exacerbated Antitumor Immune Response. Cancer research, 2020 Jul 1;80(13):2914-2926. doi: 10.1158/0008-5472.CAN-19-3584. IF 9.727
Thureau S, Faivre JC, Assaker R, Biver E, Confavreux CB, Debiais F, Duterque-coquillaud M, Giammaril F, Heymann D, Lecouvet FE, Morardet I, Paycha F, Body JJ, Vieillard MH. Adapting palliative radiation therapy for bone metastases during the Covid-19 pandemic: GEMO position paper. Journal of bone oncology, 2020 Jun; 22: 100291. doi: 10.1016/j.jbo.2020.100291. IF 2.870
Cortot AB, Audigier-Valette C, Molinier O, Le Moulec S, Barlesi F, Zalcman G, Dumont P, Pouessel D, Poulet C, Fontaine-Delaruelle C, Hiret S, Dixmier A, Renault PA, Becht C, Raffy O, Dayen C, Mazieres J, Pichon E, Langlais A, Morin F, Moro-Sibilot D, Besse B. Weekly paclitaxel plus bevacizumab versus docetaxel as second- or third-line treatment in advanced non-squamous non-small-cell lung cancer: Results of the IFCT-1103 ULTIMATE study. Eur J Cancer. 2020 May;131:27-36. IF 7.275
Marquette CH, Boutros J, Benzaquen J, Ferreira M, Pastre J, Pison C, Padovani B, Bettayeb F, Fallet V, Guibert N, Basille D, Ilie M, Hofman V, Hofman P; AIR project Study Group. Circulating tumour cells as a potential biomarker for lung cancer screening: a prospective cohort study. Lancet Respir Med. 2020 Jul;8(7):709-716. doi: 10.1016/S2213-2600(20)30081-3. PMID: 32649919. IF 15.780
Domblides C, Leroy K, Monnet I, Mazières J, Barlesi F, Gounant V, Baldacci S, Mennecier B, Toffart AC, Audigier-Valette C, Doucet L, Giroux-Leprieur E, Guisier F, Ricordel C, Molinier O, Perol M, Pichon E, Robinet G, Templement-Grangerat D, Ruppert AM, Rabbe N, Antoine M, Wislez M. Efficacy of Immune Checkpoint Inhibitors in Lung Sarcomatoid Carcinoma. J Thorac Oncol. 2020 May;15(5):860-866. doi: 10.1016/j.jtho.2020.01.014. Epub 2020 Jan 25. PMID: 31991225. IF 13.357
Jamme P, Fernandes M, Copin MC, Descarpentries C, Escande F, Morabito A, Grégoire V, Jamme M, Baldacci S, Tulasne D, Kherrouche Z* and Cortot AB*. Alterations in the PI3K pathway drive resistance to MET inhibitors in NSCLC harboring MET exon 14 skipping mutations. Journal of Thoracic Oncology. 2020 May;15(5):741. IF 13.357
Trzaska C, Amand S, Bailly C, Leroy C, Marchand V, Duvernois-Berthet E, Saliou JM, Benhabiles H, Werkmeister E, Chassat T, Guilbert R, Hannebique D, Mouray A, Copin MC, Moreau PA, Adriaenssens E, Kulozik A, Westhof E, Tulasne D, Motorin Y, Rebuffat S and Lejeune F. 2,6-Diaminopurine as a highly potent corrector of UGA nonsense mutations. Nat Commun. 2020 Mar 20;11(1):1509. doi: 10.1038/s41467-020-15140-z. IF 12.121
Turpin A, Duterque−coquillaud M, Vieillard MH. Bone metastasis: current state of play. Translational oncology. 2020 feb;13(2):308-320. IF 3.138
Baldacci S, Figeac M, Antoine M, Descarpentries C, Kherrouche Z, Jamme P, Copin MC, Tulasne D, Nanni I, Beau-Faller M, Melaabi S, Levallet G, Quoix E, Moro-Sibilot D, Friard S, Missy P, Barlesi F, Cadranel J, and Cortot AB. High MET Overexpression Does Not Predict the presence of MET exon 14 Splice Mutations in NSCLC: Results From the IFCT PREDICT.amm study. Journal of Thoracic Oncology. 2020 Jan;15(1):120-124. IF 13.357
Bennouna J, Girard N, Audigier-Valette C, le Thuaut A, Gervais R, Masson P, Marcq M, Molinier O, Cortot A, Debieuvre D, Cadranel J, Lena H, Moro-Sibilot D, Chouaid C, Mennecier B, Urban T, Sagan C, Perrier L, Barlesi F, Denis MG.Phase II Study Evaluating the Mechanisms of Resistance on Tumor Tissue and Liquid Biopsy in Patients With EGFR-mutated Non-pretreated Advanced Lung Cancer Receiving Osimertinib Until and Beyond Radiologic Progression: The MELROSE Trial. Clin Lung Cancer. 2020 Jan;21(1):e10-e14. IF 4.117
Besse B, Garrido P, Cortot AB, Johnson M, Murakami H, Gazzah A, Gil M, Bennouna J. Efficacy and safety of necitumumab and pembrolizumab combination therapy in patients with Stage IV non-small cell lung cancer. Lung Cancer. 2020 Apr;142:63-69. IF 4.599
Mazieres J, Barlesi F, Rouquette I, Molinier O, Besse B, Monnet I, Audigier-Valette C, Toffart AC, Renault PA, Fraboulet S, Hiret S, Mennecier B, Debieuvre D, Westeel V, Masson P, Madroszyk-Flandin A, Pichon E, Cortot AB, Amour E, Morin F, Zalcman G, Moro-Sibilot D, Souquet PJ. Randomized Phase II Trial Evaluating Treatment with EGFR-TKI Associated with Antiestrogen in Women with Nonsquamous Advanced-Stage NSCLC: IFCT-1003 LADIE Trial. Clin Cancer Res. 2020 Mar 6. IF 10.107
Duplaquet L, Leroy C, Vinchent A, Paget S, Lefebvre J, Vanden Abeele F, Lancel S, Giffard F, Bidaux G, Heliot L, Poulain L, Furlan A and Tulasne D. Control of cell death/survival balance by the MET dependence receptor. eLife. 2020 Feb 24;9:e50041. doi: 10.7554/eLife.50041. IF 7.551
2019
Duplaquet L, Figeac M, Leprêtre F, Frandemiche C, Villenet C, Sebda S, Sarafan-Vasseur N, Bénozène M, Vinchent A, Goormachtigh G, Wicquart W, Rousseau R, Beaussire L, Truant S, Michel P, Sabourin JC, Galateau F, Copin MC, Zalcman G, De Launoit Y, Fafeur V and Tulasne D. Functional analysis of somatic mutations affecting receptor tyrosine kinase family in metastatic colorectal cancer. Mol Cancer Ther. 2019 Mar 29. pii: molcanther.0582.2018. IF 5.365
Jamme P, Descarpentries C, Gervais R, Dansin E, Wislez M, Grégoire V, Richard N, Baldacci S, Rabbe N, Kyheng M, Kherrouche Z, Escande F, Copin MC, Cortot AB. Relevance of Detection of Mechanisms of Resistance to ALK Inhibitors in ALK-Rearranged NSCLC in Routine Practice. Clin Lung Cancer. 2019 Jul;20(4):297-304. IF 4.117
Turpin A, Duterque-Coquillaud M, Vieillard MH. Bone Metastasis: Current State of Play. Transl Oncol. 2019 Dec 23;13(2):308-320. Review. IF 3.138
Fernandes M, Duplaquet L, Tulasne D. Proteolytic cleavages of MET: the divide-and-conquer strategy of a receptor tyrosine kinase. BMB Rep. 2019 Apr;52(4):239-249. Review. IF 3.085
El Amrani M, Corfiotti F, Corvaisier M, Vasseur R, Fulbert M, Skrzypczyk C, Deshorgues AC, Gnemmi V, Tulasne D, Lahdaoui F, Vincent A, Pruvot FR, Van Seuningen I, Huet G, Truant S. Gemcitabine-induced epithelial-mesenchymal transition-like changes sustain chemoresistance of pancreatic cancer cells of mesenchymal-like phenotype. Mol Carcinog. 2019 Aug 2. IF 3.411
Gnangnon B, Fréville A, Cailliau K, Leroy C, De Witte C, Tulasne D, Martoriarti A, Jung V, Guerrera IC, Marion S, Khalife J, Pierrot C. Plasmodium pseudo-Tyrosine Kinase-like binds PP1 and SERA5 and is exported to host erythrocytes. Sci Rep. 2019 May 31;9(1):8120. IF 4.122
Vargas G, Bouchet M, Bouazza L, Reboul P, Boyault C, Gervais M, Kan C, Benetollo C, Brevet M, Croset M, Mazel M, Cayrefourcq L, Geraci S, Vacher S, Pantano F, Filipits M, Driouch K, Bieche I, Gnant M, Jacot W, Aubin JE, Duterque-Coquillaud M, Alix-Panabières C, Clézardin P, Bonnelye E. ERRα promotes breast cancer cell dissemination to bone by increasing RANK expression in primary breast tumors. Oncogene. 2019 Feb;38(7):950-964. IF 6.634
Pasquier D, Le Deley M, Tresch E, Cormier L, Duterque M, Nenan S, Lartigau E. GETUG-AFU 31: a phase I/II multicentre study evaluating the safety and efficacy of salvage stereotactic radiation in patients with intraprostatic tumour recurrence after external radiation therapy-study protocol. BMJ Open. 2019 Aug 2;9(8):e026666. IF 2.376
2018
[Delliaux C, Tian TV], Bouchet M, Fradet A, Vanpouille N, Flourens A, Deplus R, Villers A, Leroy X, Clézardin P, de Launoit Y, Bonnelye E, Duterque-Coquillaud M. TMPRSS2:ERG gene fusion expression regulates bone markers and enhances the osteoblastic phenotype of prostate cancer bone metastases. Cancer Lett. 2018 Dec 1;438:32-43. IF 6.508
Dumortier M, Ladam F, Damour I, Vacher S, Bieche I, Marchand N, DeLaunoit Y, Tulasne D and Chotteau-Lelievre A. ETV4 transcription factor and MMP13 metalloprotease are interplaying actors of breast tumorigenesis. Breast Cancer Res. 2018 Jul 11;20(1):73. IF 6.3
Descarpentries C, Leprêtre F, Escande F, Kherrouche Z, Figeac M, Sebda S, Baldacci S, Grégoire V, Jamme P, Copin MC, Tulasne D and Cortot AB. Optimization of routine testing for MET exon 14 splice site mutations in non-small cell lung cancer patients. Journal of Thoracic Oncology, 2018 Sep 6. pii: S1556-0864(18)33038-7. IF 10.336
[Baldacci S, Kherrouche Z], Cockenpot V, Stoven L, , Copin M.C, Werkmeister E, Marchand N, Maéva Kyheng, Tulasne D and. Cortot A.B. MET amplification increases the metastatic spread of EGFR-mutated NSCLC. Lung Cancer, 2018 (125)57-67. IF 4.486
Mekki S.M, Mougel A, Vinchent A, Paquet C, Copin M.C, Leroy L, Kherrouche Z, Bonte J.P, Melnyk O, Vicogne J. and Tulasne D. Hypoxia leads to decreased autophosphorylation of the MET receptor and promotes its resistance to tyrosine kinase inhibitors. Oncotarget. 2018 Jun 5;9(43):27039-27058. IF 5.168
Duplaquet L, Kherrouche Z, Baldacci S, Jamme P, Cortot AB, Copin MC and Tulasne D. The multiple paths towards MET receptor addiction in cancer. Oncogene. 2018 Jun; 37(24):3200-3215. Review. IF 7.519
Baldacci S, Kherrouche Z, Descarpentries C, Wislez M, Dansin E, Furlan A, Tulasne D, Cortot AB. MET exon 14 splicing sites mutations: A new therapeutic opportunity in lung cancer. Rev Mal Respir. 2018 Aug 31. pii: S0761-8425(18)30200-6. Review. IF 0,576
Hasne J, Hague F, Rodat-despoix L, Geerts D, Leroy C, Tulasne D, Ouadid-Ahidouh H and Kischpel P. Orai3 calcium channel and resistance to chemotherapy in breast cancer cells: the p53 connection. Cell Death Differ. 2018 Jan 11. IF 8.339
A Bokhari, V Jonchere, A Lagrange, R Bertrand, M Svrcek, L Marisa, O Buhard, M Greene, A Demidova, J Jia, E Adriaenssens, T Chassat, DS Biard, JF Flejou, F Lejeune, A Duval, A Collura. Targeting nonsense-mediated mRNA decay in colorectal cancers with microsatellite instability. Oncogenesis. 2018 Sep 19; 7(9):70; IF 5.995
2017
Deplus R, Delliaux C, Marchand N, Flourens A, Vanpouille N, Leroy X, de Launoit Y, Duterque-Coquillaud M. TMPRSS2-ERG fusion promotes prostate cancer metastases in bone. Oncotarget. 2017 Feb 14;8(7):11827-11840. IF 5.168
Jia J, Gonzalez-Hilarion S, Werkmeister E, Lafont F, Grunert D.C., Tulasne D and. Lejeune F. PTC readthrough in human cells occurs in novel cytoplasmic foci and requires UPF proteins. J Cell Sci. 2017 Jul 25. pii: jcs.198176. IF 4.431
Benhabiles H, Gonzalez-Hilarion S, Amand S, Bailly C, Prévotat A, Reix P, Hubert D, Adriaenssens E, Rebuffat S, Tulasne D, Lejeune F. Optimized approach for the identification of highly efficient correctors of nonsense mutations in human diseases. PLoS One. 2017 Nov 13;12(11):e0187930. IF 2.806
Baldacci S, Mazieres J, Tomasini P, Girard N, Guisier F, Audigier-Valette C, Monnet I, Wislez M, Pérol M, Dô P, Dansin E, Leduc C, Leprieur EG, Moro-Sibilot D, Tulasne D, Kherrouche Z, Labreuche J, Cortot AB. Outcome of EGFR-mutated NSCLC patients with MET-driven resistance to EGFR tyrosine kinase inhibitors. Oncotarget. 2017 Oct 9;8(62):105103-105114. IF 5.168
Lapère C, Cortot A.B, Grégoire V, Cockenpot V, Tulasne D, and Copin M.C. Preferential localization of MET expression at the invasion front and in spreading cells through alveolar spaces in non-small-cell lung carcinomas. Am J Surg Pathol. 2017 Mar;41(3):414-422. IF 5.363
Montagne R, Baranzelli A, Muharram G, Leroy C, Debreuck N, Kherrouche Z, Lemière A, Cortot AB and Tulasne D. Calpain cleavage of the Met receptor variant R970C generates fragment promoting epithelial cells scattering. Oncotarget. 2017 Jan 4. IF 5.168
Cortot A.B, Kherrouche Z, Descarpentries C, Wislez M, Baldacci S, Furlan A and Tulasne D. Exon 14 deleted MET receptor as a new biomarker and target in cancers. J Natl Cancer Inst. 2017 May 1;109(5). Review. IF 12,5
Lejeune F. Nonsense-mediated mRNA decay at the crossroads of many cellular pathways. BMB Rep. 2017 Apr;50(4):175-185. Review IF 3.089
Thèses en cours
- TELLIER Rémi ; D2 sous la direction de David Tulasne.
- GUERIN Célia ; D3 sous la direction de David Tulasne.
- CAROUGE Elisa ; D3 sous la direction de Anne Chotteau-Lelièvre.
Thèses soutenues
- HANTHUTE-GHESQUIER Aline (Soutenue le 06 Janvier 2023) sous la direction de : Martine DUTERQUE-COQUILLAUD.
- OLIVIER Jonathan (Soutenue le 28 Juin 2021) sous la direction de : Arnaud VILLERS.
- PALMA Martine (Soutenue le 20 Octobre 2021) sous la direction de : Fabrice LEJEUNE
- FERNANDES Marie (Soutenue le 1er Avril 2021) sous la direction de : David TULASNE.
- TURPIN Anthony (Soutenue le 30 Mars 2021) sous la direction de : Martine DUTERQUE-COQUILLAUD.
- JAMME Philippe (Soutenue le 25 Janvier 2020) sous la direction de : Alexis CORTOT et Zoulika KHERROUCHE
- DUPLAQUET Leslie (Sout. en 2018) sous le direction de : David TULASNE
- DELLIAUX Carine (sout. en 2017) sous la direction de : Martine DUTERQUE-COQUILLAUD
- DUMORTIER Mandy (sout. en 2017) sous la direction de : Anne CHOTTEAU
- BENHABILES Hana (Sout. en 2017) sous le direction de : Fabrice LEJEUNE
- BALDACCi Simon (Sout. en 2017) sous la direction de : Alexis CORTOT et Zoulika KHERROUCHE
- SIMONNEAU Claire (Sout. en 2015) sous la direction de : David TULASNE et Jérôme VICOGNE
- JIA Jieshuang (Sout. en 2015) sous la direction de : Fabrice LEJEUNE